抽象的
背景
尽管广泛的培育努力正在甘蔗(甘蔗L.),平均产量远远低于理论潜力。分蘖是甘蔗产量的重要组成部分,然而,分蘖发展的分子机制仍然难以捉摸。甘蔗的有限基因组数据,特别是由于其复杂和大的基因组,妨碍了深入的分子研究。
结果
本研究基于PacBio Iso-Seq技术,从发育中的叶片和分蘖芽样本中获取全长转录组。此外,我们对甘蔗分蘖芽T0、T1和T2三个发育阶段的样本进行rna测序,以揭示参与分蘖发育的关键基因和生物学通路。在甘蔗叶片和分蘖芽样本中分别鉴定出30360个和2088个优质非冗余亚型,分别代表甘蔗的41,109个独特亚型。同样,我们在叶片和分蘖芽样本中分别鉴定了1063个和1037个选择性剪接事件。我们预测编码序列的存在40,343个亚型,其中98%的成功注释。与之前甘蔗的FL转录组比较发现2963个未报道的亚型。此外,我们对来自11700个转录本的14946个SSRs和310个lncrna进行了特征分析。通过将RNA-seq与FL转录组整合,在T1vsT0和T2vsT0中分别鉴定出468和57个差异表达基因(DEG)。几个丙酮酸磷酸二激酶和磷酸烯醇式丙酮酸羧化酶基因的强上调表明碳固定和蛋白质合成增强,促进分蘖生长。同样,亚油酸代谢途径中亚油酸9s -脂氧合酶和脂氧合酶基因的上调表明参与分蘖生长发育的关键氧脂素的合成增多。
结论
总之,我们丰富了甘蔗的基因组数据,并提供了操纵分蘖形成和发育的候选基因,以提高甘蔗产量。
背景
甘蔗(甘蔗是禾本科重要的经济作物。它生长在热带和亚热带地区,是世界蔗糖的主要来源[1].生物燃料,纤维,肥料和其他几种副产品也来自甘蔗产量[2].据估计,超过4500万农民参与全球甘蔗产量,2019年生产近20亿吨甘蔗[1].尽管养殖广泛,但全球甘蔗平均产量(84吨/公顷)远远低于理论潜力(384 T / HA),因此,仍然需要相当大的努力来增加作物生产力[3.].
甘蔗可以收获多年,而且植物主要由茎秆组成,这些茎秆来自分蘖。类似于其他草,单一甘蔗植物可以产生多个茎[4].分蘖是甘蔗侧芽的萌动,随后发育成成熟的茎,是甘蔗产量的重要组成部分。阐明分蘖发育的分子机制对甘蔗生产具有重要意义。
分蘖早期催化了研究人员的关注。在水稻中,基因的克隆和功能鉴定MOC1型标志着分蘖调控机制的突破[5].后来,几个基因如氧化钼/Tab1./SRT1型,LAX1,LAX2,FON1.,SLR1.和TAD1.据报道,据报道协调互动MOC1型用于稻米的分蘖形成和发育[5,6,7,8,9,10,11,12,13].据报道了这一点TAD27-B.基因通过调控独脚金内酯含量来控制六倍体小麦的分蘖数[14].在玉米中,复杂的基因监管网络涉及TB1.,gt1型,tru1,Sugary1.和锡1控制分蘖发展[15.,16.,17.,18.,19.].总之,在基因组水平,分蘖是在基因组水平下,通过对各种生物途径中涉及的许多基因的表达的细化强调的多粒状性状,例如细胞周期,生长和发育和植物激素信号传导[20.,21.].在甘蔗中,到目前为止,没有记录在甘蔗中没有具体的基因控制分蘖开发。更重要的是,甘蔗在甘蔗中的分子机制仍然难以捉摸。
基因组辅助育种已成为作物改良的革命性策略,甘蔗改良潜力巨大[22.].然而,它需要高质量的基因组资源,例如完全基因组序列,重新排序和基因表达数据。甘蔗现代品种(Saccharum SPP.,2n = 100-120)是具有大(〜10 gb)和高度复杂的基因组的杂多肽[23.].已经启动了若干项目来产生甘蔗基因组学资源,特别是开发高质量参考基因组。这导致了一些基因组序列的释放,其中大多数是低质量的、碎片化的和不完整的[24.,25.,26.,27.,28.,29.].此外,在甘蔗中产生了基于下一代RNA-SEQ技术的几个转录组数据,为功能研究提供了重要的基因储存器[3.].最近,PacBio (Pacific Biosciences of California, Menlo Park, CA, USA)开发的单分子实时测序(SMRT)等第三代测序技术被用于转录组研究[30.].SMRT产生高质量的全长(FL)转录本,并促进了同源型和重复区域的基因组鉴定。对于没有良好参考基因组序列的物种,SMRT可以通过重构编码基因组进一步丰富现有的基因组资源[31.,32.].
本研究旨在重建甘蔗FL转录组,探讨分蘖发育的分子基础。为了实现这一目标,我们从甘蔗幼苗叶片和腋芽组织发育过程中制备了两个单独的PacBio Iso-Seq文库,以重建和表征FL转录组。此外,我们对甘蔗分蘖芽三个发育阶段的组织进行了rna测序,以揭示分蘖发育的差异表达基因和生物学通路。
结果
的构造与注释美国officinarum全长转录组
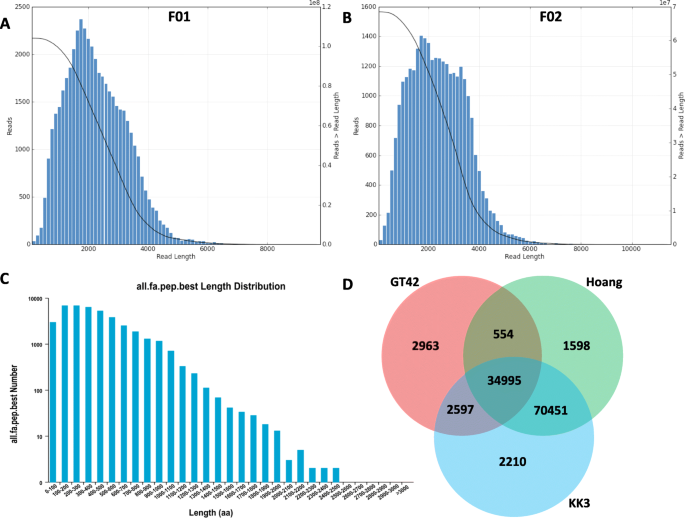
我们为叶样品(F01)和分蘖芽样品(F02)构建了两个SMRT文库。将文库分别测序3个细胞,分别产生19,7和23,63GB的F01和F02的清洁数据。在F01和F02中共鉴定了总共570,055和357,140ccs,并基于5'底漆,3'底漆和Poly(A)尾部的存在(表1).转录物长度的分布范围为150-8000和200至100,000bp,分别在f01和f02中(图。1a, b)。经过RNA-Seq reads的抛光、聚类和FL转录本的多路分解,F01和F02中分别鉴定出30,360和20,088个高质量的非冗余FLNC(表2).我们在F01和F02中分别发现了1063和1037个AS事件(表S1;S.2).通过合并来自两个库的FLNC转录物列表,我们确定了41,109个独特的FLNC成绩单美国officinarum跨越91,227,518 bp。在FL转录组中预测总共40,343个CDS,其长度范围为100至2500bp(图。1C)。使用八个不同的公共数据库进行FLNC转录物的功能注释,并且在至少一个数据库中成功注释了39,581份转录物(表3.;表S.3.).这些注释的转录本被归为3640个基因家族(表S4),其中PK基因1166个,TF基因1134个,TR基因324个(表S5).
我们将本研究中获得的独特FLNC与甘蔗中已有的FL转录组测序进行了比较[31.,32.].我们观察到,本研究鉴定的FLNC有85%在其他甘蔗品种中是保守的,而2963 FLNC与之前的甘蔗Iso-Seq研究不匹配(图。1d)。
SSRS和LNCRNA的特征
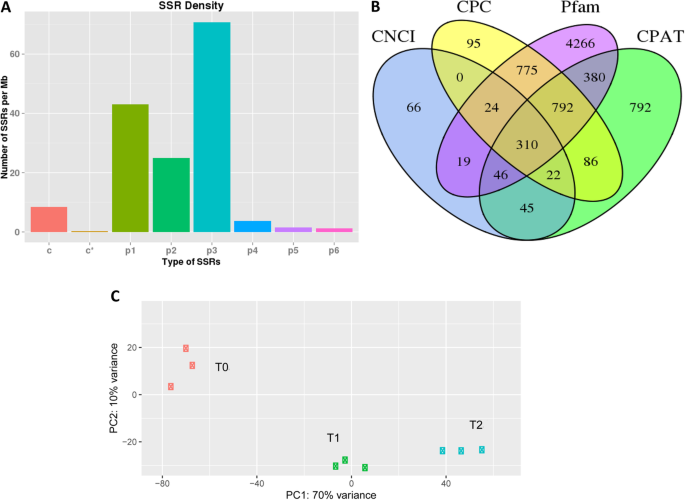
我们还检测了fll转录组中SSRs和lncrna的存在美国officinarum.我们在11,700名转录物中识别了14,946个SSR,由三核苷酸和单核苷酸SSR类型主导(图。2一种;表S.6).共有2535个基因含有超过1SSR和879种复合SSRS。关于LNCRNA,我们在用于LNCRNA预测的四种工具中检测到310 LNCRNAS(CPC,CNCI,PFAM和CPAT)中保存(图。2B;表S.7).
短阅读转录组
以Illumina-HiSeq-tenx平台为基础,从T0、T1和T2三个生物复制的分蘖芽样品中提取RNA,用于RNA-seq分析。总共有9个RNA被测序,产生超过5亿个原始短读(表S)8). 在过滤掉低质量的读取之后,我们得到了~ 79 Gb干净数据表示~ 99%的原始数据。平均而言,干净的阅读有一个Q30分~ 93%和GC含量~ 54%,显示了高质量的测序数据。干净的读取被映射到美国officinarum使用FPKM方法估计F1转录组和基因表达。我们根据FPKM数据进行了一个主要成分分析(PCA),以评估生物重复和样品的聚类模式。如图1所示。2C,所有的生物重复聚在一起,表明它们之间高度相关。此外,我们还观察到三个生长期分蘖芽的分离明显。这些结果表明,生长阶段对植物的生长有显著影响美国officinarum分蘖芽转录组。
在生长阶段的分蘖芽组织中的差异表达基因(DEG)
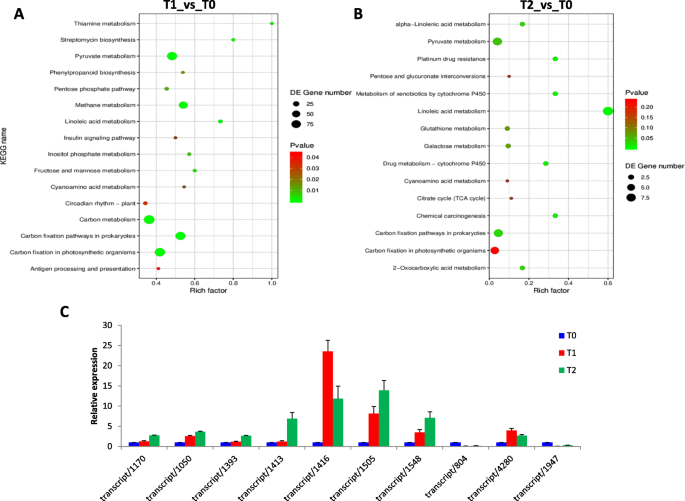
将分蘖芽T0位点的基因表达谱与T1、T2位点的基因表达谱进行比较,鉴定分蘖芽的基因表达特征。我们在T1vsT0中鉴定出468个DEGs(369个上调,99个下调),而在T2vsT0中只有57个上调,17个下调。对T1vsT0中DEGs的KEGG富集分析显示,固碳途径和丙酮酸代谢是最富集的生物途径(图1)。3.a).比较T2vsT0,我们发现固碳途径、亚油酸代谢途径和丙酮酸代谢途径是KEGG途径中最丰富的途径(图2)。3.b)。
我们随机选取10个deg,用qRT-PCR方法定量他们的转录水平(表S9).基因施作为内源性控制基因表达正常化。结果显示,与T0相比,所有所选基因在T1和T2时的转录水平都发生了显著的改变,这与RNA-seq数据中观察到的结果相似(图1)。3.C)。这些观察结果表明该研究中进行的DEG分析的可靠性。
碳固定途径
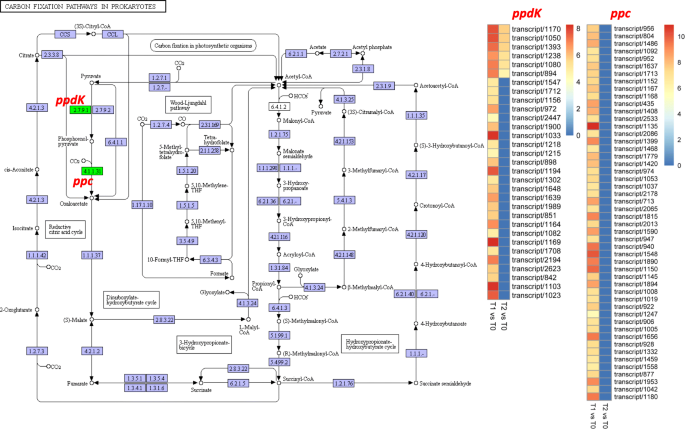
植物中的碳固定是无机碳转化为有机化合物的过程。在该研究中,筛选出81℃映射到碳固定途径。有趣的是,所有这些可作为丙酮酸磷酸盐二基因酶被注释(PPDK.)和磷酸烯醇式丙酮酸羧化酶(PPC.).这ppdks参与将丙酮酸转化为磷烯醇丙酮酸,随后转化为草酸乙酸盐PPC(图。4).然后将草酸乙酸酯还原成苹果酸,这代表了钙氏循环的输入。所有31ppdks与T0相比,T1和T2时的表达明显上调。同样,50PPC与T0相比,鉴定在T1时全部调节。总的来说,这些结果表明美国officinarum分蘖发育是强调了一个强有力的合成草酰乙酸。
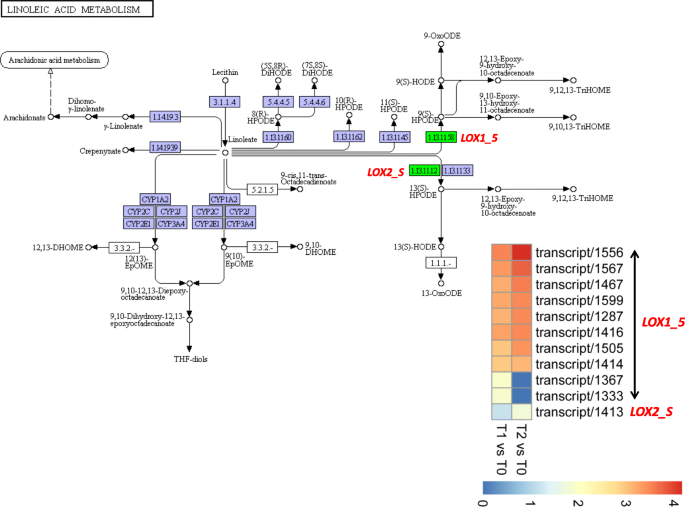
亚油酸的新陈代谢
将11个次数映射到亚油酸代谢途径,包括10个LinoLeate 9S-脂氧合酶(lox1_5.)和一种脂氧基酶(LOX2_S) (图。5).lox1_5.和LOX2_S将LinoLeate转换为(9S)-HPode和(13S)-HPode,转化为称为奥氧基吡啶的大类氧化聚苯甲酸脂肪酸[33.].与T0相比,T2和T1中这些基因的上调表明了氧化哌汀的高合成的机制,这可能是至关重要的美国officinarum分蘖成长。
转录因子
植物转录因子(TFS)规范了许多对植物生长和发展重要的生理程序。在这项研究中,我们检测到九次编码属于五个不同家庭的TFS(表4).在T1和T0之间差异表达了大多数TF,数据显示它们被上调。只有基因抄本/1947在T2VST0下调了。我们预测这些TFS在调节所涉及的结构基因方面发挥重要作用美国officinarum舵柄的发展。
讨论
Souza等人最近公布了甘蔗最大的基因组数据。28.].全长4.26 Gb,约占估计基因组大小的30%,预测了373869个基因模型。因此,很明显,在甘蔗中仍有很大比例的基因模型未被报道。此外,众所周知,超过95%的基因经历了选择性剪接事件,导致每个基因有多个同型[34.].解入甘蔗转录组中同种型的景观将提供一种有用的资源,用于改善各种植物种类所采用的基因模型预测和注释,例如小麦,玉米,韧皮和高粱[35.,36.,37.,38.].在这项研究中,我们使用PACBIO ISO-SEQ从两种甘蔗组织产生全长转录组。在叶片和分蘖组织中,总共获得了总共30,360和20,088个FLNC转录物。两种组织中FLNC转录物数量的变化突出了甘蔗中几种基因的组织特异性表达模式,这可能与重要的农艺性状相关,从而为繁殖计划提供新的标记工具[32.].与甘蔗中之前的FL转录组测序相比[31.,32.]我们得到了FLNC转录本的最低数目。这可以解释为几个因素,如组织取样,基因型,生长阶段,甚至PacBio转录组测序和数据处理程序。例如,Hoang等人[31.],在22种甘蔗品种的不同发育阶段收集来自Internode,叶子和根的RNA。他们报告了107,598个独特的成绩单,这比本研究中鉴定的FLNC转录物的数量多。尽管如此,我们在本研究中获得了最多的特定FLNC转录物。值得注意的是,在甘蔗中没有腋生分蘖芽组织转录组的先前没有报道,因此我们的结果为改善甘蔗的基因组注释提供了新的有用转录物目录。
除了有利于恢复全长转录本,PacBio Iso-Seq允许识别lncrna和重复序列,如SSRs [39.].越来越多的证据表明,lncrna是植物基因表达和基因组稳定性的关键调控因子,参与春化、育性、光形态建成、磷酸盐稳态、蛋白质重定位、染色质环动态调节等功能[40,41.].我们在这里报道了310 LNCRNA,具有高信心,这将是进一步照亮甘蔗的复杂生物学的乐器。SSRS是植物中使用的最广泛的分子标记,这归功于它们的相对丰富,共同主导,高多态性,容易和低成本的方法[42.].重要的是,已经证明了基于转录的SSRS与功能基因相关,因此它们可以用于研究它们与表型变异的关联,并且侧翼序列更容易在近距离或遥远的物种之间保守,使其用作标记比较映射更容易[43.].目前,有有限数量的甘蔗特异性SSR标记[44.因此,我们在本研究中检测到的14,946个SSRs有待验证和筛选,将对基因分型和遗传多样性研究非常有用。
通过整合三个发育阶段的分蘖芽的RNA-seq数据和我们的FL转录组,我们研究了与甘蔗幼苗分蘖发育相关的DEGs和富集途径。与T2和T0时相比,T1和T0时获得的DEGs数量更多,表明在茎蘖芽萌发过程中发生了主要的转录组调整美国officinarum幼苗。在分蘖开发期间发现了两种主要的生物途径美国officinarum幼苗:固碳和亚油酸代谢途径。
固碳是CO2掺入有机化合物中[45.],用于将能量存储和作为其他重要植物生物分子的构建块。众所周知,增强碳固定是改善植物生长和生物质积累的可行方法[46.,47.,48.].在甘蔗幼苗的分蘖开发期间,两个关键基因家族在碳固定途径中强烈调节:丙酮酸磷酸酯直立通酶(PPDK.)和磷酸烯醇式丙酮酸羧化酶(PPC.).在使用c的甘蔗等物种中4光合作用,这两种酶的作用导致合成草酸乙酸盐,其降至苹果酸盐,作为钙氏循环的输入[49.].转基因植物overexpressingPPDK.或PPC.显示出改善的碳同化和生长[50.,51.,52.,53.,54.].总的来说,我们推断出PPC.和PPDK.增加4-碳羧酸的供应,提供高碳骨架以维持高氨基酸和蛋白质合成,负责甘蔗幼苗的代谢过程增加和分蘖生长。
植物脂氧基酶(熏鲑鱼)将多不饱和脂肪酸如亚麻酸和亚油酸氧化成脂肪酸氢过氧化物,其转化为奥氧基吡啶[33.].氧脂素参与植物的生长发育等多种生理过程。例如,Kolomiets等人[55.]证明了Potlx-1在马铃薯中控制块茎的生长和发育,可能是通过启动在块茎形成过程中调节细胞生长的氧脂素的合成。在拟南芥中,已经证明9-熏鲑鱼侧根发育的调控途径[56.].类似地,lox3-4与野生型相比,敲除玉米突变体显示出根长度和植物高度,表明这一点ZMLOX3.正常植物开发Gao等人需要。[57.].强烈上调熏鲑鱼本研究中涉及的基因提示了与甘蔗分蘖生长发育有关的关键氧脂素的高合成机制。茉莉酸(JA)是一种氧脂素,多项研究表明茉莉酸的含量影响草的分蘖[58.,59.].未来的研究应该调查甘蔗耕作芽样品的培养,澄清JA或其他类型的奥氧化物促进分蘖发育。
结论
在这项研究中,我们通过从显影叶和分蘖芽组织产生和表征全长转录组,富集甘蔗中可用的基因组数据。鉴定的新型转录物将有助于甘蔗的基因组注释和基因模型预测有用。通过将RNA-SEQ数据与腋生分蘖芽组织的整合,我们鉴定了在分蘖发育过程中差异表达的碳固定和亚油酸途径的重要基因。对这些候选基因的进一步深入调查将提供用于控制甘蔗的耕作过度和生产率的前景。
方法
植物材料
在这项研究中,美国officinarumL.栽培品种Guitang 42(GT42)用作植物材料。它是一种优良的品种,具有高产,高糖,耐腐蚀性,适用于中国广西农业科学院甘蔗研究所的机械化生产[60.].植物材料来源于中国广西农业科学院甘蔗研究所。该材料的正式鉴定由本研究的通讯作者邱立航教授进行。植物材料的凭证没有存入基因库。取健康茎,切成单芽段,用水冲洗,浸泡30 min。将30个只含1芽的茎作为繁殖体,置于24 × 19.5 × 26.5 cm的盆中,每盆3个繁殖体,每盆灌满11 kg泥炭营养土,在广西农业科学院甘蔗研究所试验田(纬度22.85,经度108.24,海拔50.17 m)的大棚内覆盖塑料薄膜,温室内甘蔗生长的小气候条件如下:平均温度为36.52±0.76℃,平均相对湿度为75.66±2.4%,平均光强为1884.1±39.25 μmol m- 2 S- 1.当芽体从土壤中发芽时,将塑料薄膜除去,并在每个花盆中喷洒水,以确保芽体的正常发育。随后,分别在不同生长阶段采集甘蔗叶片组织和分蘖芽样本,包括T0(2-3叶期;发育期),T1(4-5叶期;分蘖期开始),T2(6-7叶期;分蘖期)。对于叶片样本,从每个花盆的三种植物中收集第一个真正的叶片,混合形成生物复制。对于分蘖芽样本,T0时腋生分蘖芽开始在地下萌发。T1时,腋生分蘖芽在地下约0.5 cm - 1 cm长。T2时,腋生分蘖芽从土壤中萌发,长度约为2厘米。 At each stage, axillary tiller buds were collected from the three plants of each pot and mixed to form a biological replicate. Samples were collected in three biological replicates from plants grown in different pots and quickly frozen with liquid nitrogen and stored at − 80 °C.
图书馆施工和单分子实时(SMRT)测序
通过在三个生长阶段收集的混合叶样品和来自三个生长阶段的混合分蘖芽样品进行磨削的总RNA,分别在制造商的协议之后分开,在制造商的协议之后,构建了两个图书馆。用Agilent 2100生物分析仪(Agilent Technologies,Santa Clara,Ca,USA)和琼脂糖凝胶(1%)电泳来确定RNA的完整性。用纳米玻璃微量分光光度计(Thermo Fisher Scientific,Waltham,Ma,USA)测定RNA的纯度和浓度。通过寡核苷酸(DT)磁珠富集MRNA。然后使用Clontech Smarter PCR cDNA合成试剂盒(Clontech Laboratories,USA),富集的mRNA被逆转录成cDNA。PCR循环优化用于确定下游大规模PCR反应的最佳放大循环数。然后,优化的循环次数用于产生双链cDNA。此外,使用蓝色Pippin TM尺寸选择系统进行1-6 kB尺寸选择。然后,对下一个SMRTBELL图书馆构建进行大规模的PCR。修复CDNA以进行DNA损伤并连接到测序适配器。 The SMRTbell template was annealed to sequencing primer, bound to polymerase, and sequenced on the PacBio Sequel platform using P6-C4 chemistry with 10 h movies by Biomarker Technology Co. (Beijing, China).
PacBio长读处理
PacBio数据处理跟踪了Biomarker Technology Co.(北京,中国)建立的标准程序。使用SMRT链路V5.01pipeline对CDNA文库的原始测序读入cDNA文库的读数并聚集成转录人共识[61.].首先,将循环共识序列(CCS)读出读出Subreads BAM文件。接下来,CCS读数被分类为全长非嵌合(FL),非全长(NFL),嵌合体和基于cDNA引物和多尾信号的短读取。从分析中丢弃短读取(<50 bp)。随后,通过迭代聚类聚类全长非嵌合读取用于纠错软件以生成群集共识同种型。为提高PACBIO读取的准确性,采用了两种策略。首先,NFL读数用于通过Quivier软件抛光上述群集共识同种型[62.]获得FL抛光的高质量共识序列(精度≥90%)。接下来,使用Lortec工具(版本0.8)使用Illumina短读取的低质量同种型进一步纠正63.].然后,通过用软件CD-HIT-V4.6.7除去冗余序列来过滤最终转录组同种次序列[64.]使用0.99的标识阈值。
诠释基因和转录因子(TF)的鉴定,转录调节剂(TR)和蛋白激酶(PK)
我们使用了TransDecoder软件(https://github.com/TransDecoder/TransDecoder/wiki)对于编码序列(CD)预测。接下来,将FLNC转录物的DNA或蛋白质序列提交给各种公共数据库进行功能注释:立即组群/真核外科群(COG / KOG)数据库(http://www.ncbi.nlm.nih.gov/cog.),基因本体(GO),NCBI非冗余蛋白(NR),Swiss-Prot蛋白(Swissprot)数据库(http://www.expasy.ch/sprot.),基因和基因组(Kegg)数据库的京都百科全书(http://www.genome.jp/kegg.),基因的进化课程:非监督正非群体(Eggng)数据库(http://eggnog5.embl.de/)和pfam蛋白质(pfam)数据库(https://pfam.xfam.org/).为了鉴定植物转录因子(TFs)、转录调节因子(TRs)和蛋白激酶(PKs),将转录本提交到植物转录因子数据库(PlantTFdb:http://planttfdb.gao-lab.org/, [65.])和Itak(v.1.5,[66.])以最佳匹配结果[67.].
简单的序列重复(SSR)预测
微卫星(MISA,http://pgrc.ipk-gatersleben.de/misa//)是一套可识别七种类型的简单序列重复(SSR)的软件包:单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸和复合SSR(杂交微卫星)。我们通过以下参数筛选了长度为> 500 bp的转录本:unit_size- min_repeats: 2 - 6,3 - 5,4 - 4,5 - 4,6 - 4;化合物(max_difference_between_2_SSRs): 100。
长期非编码RNA(LNCRNA)的表征
使用四个工具来预测LNCRNA:CNCI(v.2)[68.],CPC(V.0.9-R2)[69.]),pfam蛋白质(pfam)数据库(https://pfam.xfam.org/)和cpat(v.3.0.0)[70].在本研究中,我们将四种工具之间常用检测到的lncrna作为最可能的lncrna。
替代拼接(AS)检测
随着转录物被预测到PAN描述的方法et al。[71.].所有序列都以高身份设置运行全VS-所有BLAST [72.]针对组装的FL转录组。符合所有标准的BLAST对准被认为是候选的产品作为事件:(1)序列(查询和主体)长度超过1000bp,对齐包含2个高分段对(HSP);(2)替代剪接差距超过100bp,并从3'/ 5'端位于≥100bp;(3)所有替代转录物允许5-BP重叠。
RNA-SEQ图书馆构建和排序
根据制造商的议定书,从三种生长阶段收集的三次生长阶段收集的分蘖芽中提取了高质量的RNA,根据制造商的议定书,通过Trizol®试剂(天根北京)。通过使用无RNase的DNase I(Takara,Kyoto,Japan)纯化来自不同样品的RNA,以除去基因组DNA污染。在Biomarker Technology Co.(北京,中国)建立的标准程序下进行了Illumina Hiseq Ten X平台上的cDNA图书馆建设和测序,并通过Li等人完全描述。[73.].
基因表达定量及差异表达基因(DEG)分析
原始RNA序列数据由fastQC v0.11.2处理。使用Bowtie2将成对末端清洁读数与参考FL转录组对齐[74.]默认参数。基因表达被每百万碱基对测序(FPKM)方法的转录物序列每千碱基的片段数量估计。使用DESEQ2封装进行差异表达分析。具有折叠变化≥2的基因和假发现速率(FDR)<0.05被认为是在比较分析中显着差异表达的基因(DEGS)。对Kegg的富集分析进行了次数[75.]及COG [76.途径。为了验证DEG分析,选择10个基因,基于QRT-PCR方法在三个生物学和三种技术复制后验证了它们的表达水平,并在Dossa等人的描述下进行了描述。[77.].
可用性数据和材料
RNA-SEQ数据已提交给NCBI SRA:PRJNA723212并可访问www.ncbi.nlm.nih.gov sra / PRJNA723212.
缩写
- 度:
-
差异表达基因
- FL:
-
全长
- FLNC:
-
全长非编码
- 主成分分析:
-
主要成分分析
- FPKM:
-
每百万次碎片每千碱基碎片映射
- TF:
-
转录因子
- lncrana:
-
长编码RNA
- SSR:
-
简单序列重复
参考
- 1.
FAOSTAT 2018.可访问:http://www.fao.org/faostat/en/#home.. 于2020年8月25日咨询。
- 2。
魏Y-A,Li Y-R。中国糖业的现状与趋势。糖科技。2006; 8(1):10-5。https://doi.org/10.1007/BF02943735.
- 3.
Diniz Al,Ferreira Ss,Caten Ft,Margarido Gra,Dos Santos JM,Barbosa Gv等。后基因组学时的能量甘蔗基因组资源。计算结构Biotechnol J. 2019; 10:1404-14。
- 4.
王国王,侯爵伦,休斯。手杖生长手册。纽约:美国elsevier出版公司,公司;1965年。375。
- 5。
李X,钱Q,Fu Z,王Y,Xiong G,Zeng D等人。稻米分蘖的控制。自然。2003; 422(6932):618-21。https://doi.org/10.1038/nature01518.
- 6。
廖Z,余H,段J,Yuan K,Yu C,Meng X等。SLR1抑制MOC1劣化以协调米饭中的分蘖数和植物高度。NAT Communce。2019; 10(1):2738。https://doi.org/10.1038/s41467-019-10667-2.
- 7。
acta photonica sinica, 2011, 41(5): 689 - 693 .卢卓,邵刚,熊军,焦勇,王军,刘刚,等。独角兽3,orthologWUSCHEL在水稻中,是分蘖芽形成所必需的。J. Genet Genomics. 2015; 42:71-8。
- 8。
MJOMBA F,郑Y,刘H,唐W,Hong Z,Wang F等人。Homeobox是关键的oswus.水稻分蘖发育和雌性育性的控制。G3。2016; 6:2013-21。
- 9。
Oikawa T、Kyozuka j两步调节PANICLE1松懈水稻腋生分生组织形成过程中蛋白质的积累。植物细胞。2009;21(4):1095 - 108。https://doi.org/10.1105/TPC.108.065425.
- 10.
acta photonica sinica, 2011, 38(5): 689 - 693 .邵刚,陆震,熊军,王斌,景勇,孟x,等。水稻分蘖芽形成调控因子MOC1和MOC3通过激活FON1的表达共同促进分蘖芽的生长。摩尔。2019;12(8):1090 - 102。https://doi.org/10.1016/j.molp.2019.04.008.
- 11.
田渊,张勇,Hattori S, Omae M, Shimizu-Sato S, Oikawa T,等。PANICLE2松懈大米编码一种新型核蛋白,并调节腋生分析的形成。植物细胞。2011; 23(9):3276-87。https://doi.org/10.1105/tpc.111.088765.
- 12.
Tanaka W,Ohmori Y,Ushijima T,Matsusaka H,Matsushita T,Kumamaru T等人。稻米腋生成分形成需要WUSCHEL正射分蘖缺席1.植物细胞。2015; 27(4):1173-84。https://doi.org/10.1105/tpc.15.00074.
- 13。
徐C,王Y,yu Y,段J,Liao Z,Xiong G等。通过APC / C(TAD1)降解单链体1(TAD1)调节米粉分蘖。NAT Communce。2012; 3(1):750。https://doi.org/10.1038/ncomms1743.
- 14。
赵b,吴tt,ma ss,jiang dj,bie xm,sui n等。TAD27-B.基因控制六倍体小麦的分蘖数。植物营养与肥料学报;2010;https://doi.org/10.1111/pbi.13220.
- 15.
董泽,等。理想的作物植物架构是由流苏取代上部耳朵1,直接由Teosinte支链直接靶向的BTB / POZ Ankyrin重复基因。Proc Natl Acad Sci U S A. 2017; 114(41):E8656-64。https://doi.org/10.1073/pnas.1714960114.
- 16。
Kebrom Th,Brutnell TP。分蘖在Sugary1.通过覆盖来保持甜玉米Teosinte分支机构1压制性的信号。植物信号行为研究。2015;10(12):e1078954。https://doi.org/10.1080/15592324.2015.1078954.
- 17。
[11]赵庆华,张建平,张建平,等。一个玉米驯化基因转座子的插入TB1..NAT Genet。2011; 43(11):1160-4。https://doi.org/10.1038/ng.942.
- 18。
杨建平,杨建平,王永平,等。长满草的tillers1促进玉米的顶级优势,并对草中的阴影信号作出反应。PROC NATL ACAD SCI U S A. 2011; 108(33):E506-12。https://doi.org/10.1073/pnas.1102819108.
- 19。
张X,林Z,王J,刘H,周L,钟S等锡1基因保留促进玉米耕作的功能。NAT Communce。2019; 10(1):5608。https://doi.org/10.1038/s41467-019-13425-6.
- 20。
多玛加尔斯卡·马,莱瑟·O。枝梢分枝控制中的信号整合。Nat Rev Mol细胞生物学。2011;12(4):211–21.https://doi.org/10.1038/nrm3088.
- 21。
王艳红,李建英。水稻的分枝。植物生物学杂志。2011;14(1):94-9。https://doi.org/10.1016/j.pbi.2010.11.002.
- 22。
坎德尔R,杨X,宋J,王J。甘蔗生物量改良的潜力、挑战及遗传和基因组资源。前植物科学。2018;9:151.https://doi.org/10.3389/fpls.2018.00151.
- 23。
Piperidis N,D'Hont A.甘蔗基因组结构用染色体特异性寡核苷酸探针解密。工厂J. 2020; 103(6):2039-51。https://doi.org/10.1111/tpj.14881.
- 24。
de Setta N、Monteiro Vitorello CB、Metcalfe CJ、Cruz GMQ、Del Bem LE、Vicentini R等。构建甘蔗生物技术基因组并确定进化趋势。BMC基因组学。2014;15:540.https://doi.org/10.1186/1471-2164-15-540.
- 25。
Garsmeur O,Droc G,Antonise R,Grimwood J,Potier B,Aitken K,等。甘蔗高复杂基因组的马赛克单倍体参考序列。NAT Communce。2018; 9。https://doi.org/10.1038/s41467-018-05051-5.
- 26。
Nascimento LC,Yanagui K,Jose J,Camargo Elo,Grassi MCB,Cunha CP等人。解开复杂的基因组Saccharum spontaneum.使用多倍体基因汇编程序。DNA Res。2019; 26:205-16。https://doi.org/10.1093/DNARES/DSZ001.
- 27。
Riaño-PachónDM,Mattiello L.甘蔗杂交SP80-3280的基因组测序草案。F1000res。2017; 6:861。https://doi.org/10.12688/f1000Research10.12688/F1000Research.11859.2..
- 28。
Souza GM、Van Sluys MA、Lembke CG、Lee H、Margarido GRA、Hotta CT等。多倍体甘蔗基因组373K基因空间的组装揭示了世界主要生物量作物的功能多样性库。Gigascience公司。2019;8(12):第129页。https://doi.org/10.1093/gigascience/giz129.
- 29。
张j,张x,唐h,张q,hua x,ma x等。等位基因定义的自动聚合物甘蔗的基因组Saccharum spontaneum.L. Nat Genet. 2018; 50:1565-73。https://doi.org/10.1038/s41588-018-0237-2.
- 30.
王B,Kumar V,Olson A,洁具D.恢复转录组研究:深入了解单分子转录组测序的出现。前群体。2019; 10:384。https://doi.org/10.3389/fgene.2019.00384.
- 31.
Hoang NV,Furtado A,Mason PJ,Marquardt A,Kasirajan L,Thirugnanasambandam PP等。利用全长异构体测序和短阅读测序的从头组装对高度多倍体甘蔗基因组的复杂转录组进行调查。BMC基因组学。2017;18(1):395.https://doi.org/10.1186/s12864-017-3757-8.
- 32.
Piriyapongsa J,Kaewprommal P,Vaiwsri S,Anuntakarun S,Wirojsirasak W,Punpee P等人。使用单分子长读测序揭示甘蔗品种Khon Ken3的全长转录物同种型。peerj。2018; 6:E5818。https://doi.org/10.7717/peerj.5818.
- 33.
porta h,rocha-sosa m.植物脂氧基酶。生理和分子特征。植物理性。2002; 130(1):15-21。https://doi.org/10.1104/pp.010787.
- 34.
Pan Q, Shai O, Lee LJ, Frey BJ, Blencowe BJ。通过高通量测序对人类转录组中可变剪接复杂性的深入调查。Nat麝猫。2008;40(12):1413 - 5。https://doi.org/10.1038/ng.259..
- 35.
Abdel-Ghany Se,Hamilton M,Jacobi JL,Ngam P,Devitt N,Schilkey F等人。使用单分子长读取高粱转录组的调查。NAT Communce。2016; 7(1):11706。https://doi.org/10.1038/ncomms11706..
- 36。
董立,刘立,张继,杨某,孔G,Chu Js等。单分子实时转录测序促进常见的小麦基因组注释和晶粒转录组研究。BMC基因组学。2015; 16(1):1039。https://doi.org/10.1186/s12864-015-2257-y.
- 37。
Jo Ih,Lee J,Hong Ce,Lee Dj,Bae W,Park SG等人。同种型测序提供了对Panax人参转录组的更全面的观点。基因。2017; 8(9):228。https://doi.org/10.3390/genes8090228.
- 38。
王b,tseng e,scallski m,clark ta,hon t,jiao y等。通过单分子长读取测序揭示玉米转录组的复杂性。NAT Communce。2016; 7(1):11708。https://doi.org/10.1038/ncomms11708.
- 39。
Rhoads A, Au KF。PacBio测序技术及其应用。基因组学、蛋白质组学、生物信息学。2015;13(5):278-89。https://doi.org/10.1016/j.gpb.2015.08.002.
- 40
切卡诺娃。长非编码rna及其在植物中的功能。植物生物学。2015;27:207–16.https://doi.org/10.1016/j.pbi.2015.08.003.
- 41
关键词:非编码rna,植物,生物学功能,生物信息学基因组学、蛋白质组学、生物信息学。2015;13(3):137-47。https://doi.org/10.1016/j.gpb.2015.02.003.
- 42
Dossa K,Yu J,Liao B,Cisse N,Zhang X.开发高度信息的基因组宽单序列重复标志,用于育种应用中的育种应用和网络资源的构建:Sisatbase。前植物SCI。2017; 8:1470。https://doi.org/10.3389/fpls.2017.01470.
- 43
Tulsani NJ,Hamid R,Jacob F,Umretiya Ng,Nandha Ak,Tomar Rs等。基因挖掘和SSR标志性植物的转录综合组合在香菜(Coriandrum Sativum L.)中。基因组学。2020; 112(2):1545-53。https://doi.org/10.1016/j.ygeno.2019.09.004.
- 44。
基于ssr的分子鉴定数据库的开发与整合。农学。2016;6(2):28。https://doi.org/10.3390/agronomy6020028.
- 45。
酒吧 - 偶数A,Noor E,Lewis Ne,Milo R.合成碳固定途径的设计与分析。PROC NATL ACAD SCI。2010; 107(19):8889-94。https://doi.org/10.1073/pnas.0907176107.
- 46。
即使是A。大胆的代谢设计增强植物碳固定。植物科学。2018;273:71–83.https://doi.org/10.1016/j.plantsci.2017.12.007.
- 47。
作者简介:Betti M, Bauwe H, Busch FA, Fernie AR, Keech O, Levey M等。操纵光呼吸提高植物生产力:作物改良的最新进展和展望。实验机器人。2016;67(10):2977-88。https://doi.org/10.1093/jxb/erw076.
- 48。
杜坎特特区,银PA。改善固碳途径。中国生物医学工程学报,2012;https://doi.org/10.1016/j.cbpa.2012.05.002.
- 49。
布朗Rh,Byrd GT,Bouton Jh,Bassett Cl。杂交物中杂交种的光合特性(C4样)与黄细胞线性(C3-C4)。植物理性。1993; 101:825-31。
- 50.
Giuliani R,Karki S,Covshoff S,Lin HC,Coe Ra,Koteyeva NK等。转基因玉米磷酸丙酮酸羧基酶改变叶片大气2和13有限公司2水稻交易所。Photosynth研究》2019;142(2):153 - 67。https://doi.org/10.1007/S11120-019-00655-4.
- 51.
Sen P,Ghosh S,Sarkar Sn,Chanda P,Mukherjee As,Datta K,等人。金字塔三c4水稻增产的特定基因。植物细胞组织器官培养。2017;128(1):145-60。https://doi.org/10.1007/S11240-016-1094-2.
- 52.
丙酮酸-磷酸二激酶(pyruvate-phosphate Dikinase)4-SMPPDK.)基因来自碱蓬monoica增强C的光合作用,碳同化和非生物胁迫耐受性3.高CO下植物2条件。植物科学与技术,2000,21(1):1 - 5。https://doi.org/10.3389/fpls.2020.00345.
- 53.
Yadav S,Mishra A. C的异位表达4光合途径基因可提高碳同化,减轻对未来气候变化的应力耐受性。physiolmol biol植物。2020; 26(2):195-209。https://doi.org/10.1007/S12298-019-00751-8.
- 54.
Zhang J,Bandyopadhyay A,Sellappan K,Wang G,Xie H,Datta K,等。表征C4玉米丙酮酸正磷酸二激酶在C3.转基因水稻。生物技术学报。2010;9(2):234-42。
- 55.
Kolomiets MV,Hannapel DJ,Chen H,Tymeson M,Gladon RJ。脂氧合酶参与马铃薯块茎发育的控制。植物细胞。2001; 13(3):613-26。https://doi.org/10.1105/tpc.13.3.613.
- 56.
Vellosillo T,Martínezm,洛佩斯马,维森特j,cascónt,dolan l等。通过拟南芥中的9-脂氧合酶途径产生的奥氧化物通过特定信号级联调节横向根部发育和防御反应。植物细胞。2007; 19(3):831-46。https://doi.org/10.1105/tpc.106.046052.
- 57.
高X,Starr J,GöbelC,Engelberth J,Feussner I,Tumlinson J等。玉米9-脂氧合酶ZMLOX3对抗外防基因的发育,根特异性表达,以及对根结线虫的抵抗力。Mol植物微生物相互作用。2008; 21(1):98-109。https://doi.org/10.1094/MPMI-21-1-0098.
- 58.
刘X,李福,唐家璇,王文,张F,王g等。氢过氧化物溶酶OSHPL3耗尽茉莉酸途径的激活揭示了水稻中奥克脂途径的HPL和AOS分支之间的串扰。Plos一个。2012; 7(11):E50089。https://doi.org/10.1371/journal.pone.0050089..
- 59.
刘克,芬拉森SA。通过与覆盖叶接触来压抑高粱分蘖芽增长。植物细胞环境。2019; 42(7):2120-32。https://doi.org/10.1111/pce.13548.
- 60.
王L,Liao J,Tan F,Tang S,Huang J,Li X等。新型高产,高糖和耐植物甘蔗品种冠塘42的育种及其高产栽培技术。J南部农业。2015; 46:1361-6。
- 61
Gordon SP,Tseng E,Salamov A,Zhang J,Meng X,Zhao Z等。单分子mRNA测序揭示真菌的广泛多函数转录物。Plos一个。2015; 10(7):E0132628。https://doi.org/10.1371/journal.pone.0132628.
- 62
陈建平,陈建平,陈建平,等。非杂交,完成的微生物基因组组装从长读SMRT测序数据。Nat方法。2013;10:563-9。
- 63
Salmela L,竞争对手E.Rordec:准确,高效的长读误差校正。生物信息学。2014; 30(24):3506-14。https://doi.org/10.1093/bioinformatics/btu538.
- 64
富L,NIU B,Zhu Z,Wu S,Li W.CD-HIT:加速为聚类下一代测序数据。生物信息学。2012; 28:3150-2。
- 65
金杰,田F,杨立德,孟悉,孔L,罗杰,等。Planttfdb 4.0:朝向植物中的转录因子和监管相互作用的中心集线器。核酸RES。2017; 45(D1):D1040-5。https://doi.org/10.1093/nar/gkw982.
- 66
郑义,焦思,孙H,罗斯利·哈克,普通马,张P等人。ITAK:用于植物转录因子,转录调节剂和蛋白激酶的基因组预测和分类的程序。莫尔植物。2016; 9(12):1667-70。https://doi.org/10.1016/j.molp.2016.09.014.
- 67
杨X,夏x,张z,nong b,zeng y,wu y等。用PCAMP鉴定水稻果皮中的花青素生物合成基因。植物Biotechnol J. 2019; 17(9):1700-2。https://doi.org/10.1111/pbi.13133.
- 68。
孙璐,罗洪,卜丹,等。利用序列内在组成对蛋白质编码和长非编码转录本进行分类。核酸研究2013。https://doi.org/10.1093/nar/gkt646.
- 69。
孔璐,张勇,叶志强,等。CPC:利用序列特征和支持向量机评估转录本的蛋白质编码潜力。核酸研究2007;35(Web服务器):W345-9。
- 70。
关键词:编码潜能评估,无对齐logistic回归模型,编码潜能评估2013;41(6):e74。https://doi.org/10.1093/nar/gkt006.
- 71。
Pan L,Guo M,Jin X,Sun Z,Jiang H,Han J等人。寄生虫黄蜂的全长转录组调查及表达分析Chouioia cunea暴露在1-十二章。SCI批准。2019; 9(1):18167。https://doi.org/10.1038/s41598-019-54710-0.
- 72。
Altschul SF,Madden TL,SchäfferAA,张J,Zhang Z,Miller W等人。Papped Blast和PSI Blast:新一代蛋白质数据库搜索程序。核酸RES。1997年; 25(17):3389-402。https://doi.org/10.1093/nar/25.17.3389.
- 73.
李玲,吴华,马鑫,徐伟,梁强,詹锐,等。两个对照芒果(Mangifera indica L.)果肉中糖差异积累的转录机制。基因组学。2020;112(6):4505 - 15所示。https://doi.org/10.1016/j.ygeno.2020.07.038.
- 74.
Langmead B, Salzberg S.快速缺口读对齐与领结2。Nat方法。2012;9(4):357 - 9。https://doi.org/10.1038/nmeth.1923.
- 75.
Kanehisa M、Araki M、Goto S、Hattori M、Hirakawa M、Itoh M、Katayama T、Kawashima S、Okuda S、Tokimatsu T、Yamanishi Y。KEGG将基因组与生命和环境联系起来。核酸研究,2008年;36:D480-4。https://doi.org/10.1093/nar/gkm882..
- 76.
Tatusov RL,Galperin My,Natale da。COG数据库:蛋白质功能和演化的基因组规模分析的工具。核酸RES。2000; 28(1):33-6。https://doi.org/10.1093/nar/28.1.33.
- 77.
周锐,张涛,苏瑞,张勇,等。芝麻多种非生物胁迫响应的核心转录组(芝麻纪录L.)。int j mol sci。2019; 20(16):3930。
致谢
不适用。
资金
国家自然科学基金项目(no . 31701363, no . 31960416, no . 31360312);广西自然科学基金项目(no . 2018GXNSFAA138149, no . 2018GXNSFDA294004, no . 2016GXNSFBA380034, no . 2015GXNSFDA139011);广西科技项目(桂科AD19245080)、广西农业科学院基金项目(2020YM22、31701363、2018YM02、2018YT01)。资助者不参与研究设计、数据收集和分析、决定出版或手稿编写。
作者信息
隶属关系
贡献
H. Y.、H. Z.、H. L.和Y. F.收集植物材料并进行实验;张志忠,荣昌,田林,徐国良。L,ξ。L.和Y. L.进行了生物信息学分析;H. Y.和H. Z.撰写了论文;l.q.和j.w.设计了这项研究,指导了这项研究并提供了资金。所有作者已经阅读并批准了本手稿的最终版本。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
提交人声明他们没有利益冲突。
附加信息
出版商的注意
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
附加文件1:表S1。
识别替代拼接事件和同种型美国officinarum基于叶片样本库的全长转录组研究。表S2。识别替代拼接事件和同种型美国officinarum基于分蘖芽样品库的全长转录组。表S3。基因注释统计。表S4。基因家族统计。表S5。作为转录因子(TF),转录调节剂(TRS)和蛋白激酶(PK)注释的基因列表。表S6。描述简单序列重复在美国officinarum完整的转录组。表S7。LNCRNA的特征美国officinarum完整的转录组。LNCRNA以红色突出显示的是四个软件常用的LNCRNA。表S8。Illumina RNA-SEQ的统计数据美国officinarum分蘖芽经过三个生长阶段。表S9。用于差异表达基因的QRT-PCR验证的基因的引物序列。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用本文
燕,H.,周,H.,Luo,H.et al。中全长转录组的特征甘蔗和分子洞察分蘖发展。BMC植物BIOL.21,228(2021)。https://doi.org/10.1186/s12870-021-02989-5
收到了:
接受:
发表:
关键字
- 作物生产力
- 基因组数据
- C4植物
- 碳固定
- 亚油酸
- 基因表达