摘要
背景
豆科植物中含有较多的IVa碱性-螺旋-环-螺旋(bHLH)转录因子亚支,该亚支中的一些成员已被鉴定为皂苷生物合成调节因子。本研究旨在系统阐明豆科植物皂苷生物合成调控的多样性,并深入了解豆科植物皂苷生物合成调控的进化历史。
结果
在本研究中,我们收集了40种植物(包括豆科植物和其它植物)的IVa-bHLH亚基蛋白质序列,发现在豆科植物中有更多的IVa-bHLH亚基。我们证实了bHLH结构域、C-末端actlike结构域和外显子-内含子组织在模式豆科几乎所有IVa亚类成员中的保守性,支持了我们的分类结果。基于系统发育树的IVa亚类分类显示存在三个不同的类群。有趣的是,大多数豆科植物的IVa-bHLHs亚类都属于第1组,其中包含迄今为止鉴定的所有豆科皂甙生物合成调节因子。这些观察结果支持共同存在和豆科特有的多样化的皂苷生物合成调节。同源基因在大肠杆菌中表达的比较大豆那Medicago Truncatula.,Lotus对虾,MTTSAR1(第一个鉴定的大豆酶生物合成调节转录因子)在同一组织中不表达,表明,在重复和分歧期间,1族成员对皂苷生物合成产生了不同的表达模式和贡献。另一方面,2和3组具有更少的成员,并且它们的系统发育关系和表达模式高度保守,表明它们的活性可能会在Fabaceae上保守。
结论
该研究表明Fabaceae植物中亚洲IVA BHLHs的细分和多样化。结果对于未识别的皂苷生物合成调节剂的候选选择是有用的。此外,组2和3个成员的功能是有趣的,用于阐明Fabaceae中亚洲IVA BHLH转录因子的亚洲IVA BHLH转录因子的演变的目标。
背景
三萜皂苷是一组广泛存在于植物界的植物特化(次级)代谢物[1].在其生物合成方面已经深入研究了Triterpenes [2,转录调控[3.那4.那5.,以及生物活性[6.那7.那8.].在属的单圈子植物艾薇娜产生抗真菌皂苷,称为百叶香蛋白,用于防止服用疾病[9.].皂苷生产的Barbarea寻常魅力(芸薹科)对昆虫有拒食活性[10].Fabaceae(豆科植物)植物在结构多样化的三角形中产生,包括溶血性皂苷Medicago Truncatula.,桦木酸Lotus对虾和glycyrrhizin乌拉尔甘草,以及一种常见的大豆皂甙[11那12那13那14那15].豆科三萜胶片影响共生染色剂,如转基因M. Truncatula.含有升高的皂苷含量显示增强的染色量[16]以及l .对虾缺乏鹿皮酚(桦木酸的前体)的突变体表现出快速结瘤的表型[17].
碱性-螺旋-环-螺旋(bHLH)转录因子是植物中最大的转录因子家族之一,根据bHLH结构域和其他共享蛋白结构域的序列同源性,可分为约25个亚支[18那19].陆地植物比动物、绿藻或红藻获得了更多的bHLH基因[19],一些亚分支进化来调节植物的特化代谢[5.].亚分支IVa是这种调节的一个很好的例子,因为它代表了茉莉酸甲酯(MeJA)介导的植物代谢过程的保守转录调节[5.].三萜皂苷生物合成激活调节因子1 (MtTSAR1)上调大豆皂苷通路M. Truncatula.[20.].mttsas2和3是激活溶血皂苷积累的因子,在组织特异性上存在差异[20.那21].我们最近将GubhlH3鉴定为豆楸生物合成的阳性调节器G. Uralensis.[22[该蛋白质与MTTSAR2密切相关但不是MTTSAR1。这在Fabaceae Subclade IVA BHLHS的进化历史中发现了暗示。藜藜麦(Amaranthaceae)种子积累皂苷与类似的结构与溶血皂苷M. Truncatula.. 基因突变CqTSAR-like1(cqtsarl1.)是影响皂苷生产型和无皂苷生态型之间皂苷积累模式差异的主要因素[23].在Catharanthus roseus也叫(apocynaceae),BHLH IRIDOID合成1(CRBIS1)和CRBIS2被发现积极调节单萜类吲哚生物碱(MIAS)的虹膜侧柑橘分支的生物合成途径[24那25].有趣的是,MTTSAR和CRBIS1的功能显示通过MTTSAR的异源表达可互换C. Roseus.和CrBIS1M. Truncatula.[26].此外,皂苷和MIAs的生产通常都受到MeJA的调控[5.那21那24那27].
大量研究报道了植物中bHLH因子的全基因组鉴定和分类[18那19那28那29那30].尽管基因组拟南芥和奥雅萨苜蓿分别拥有四个及六个IVa次级成员[19]的基因组中发现了30多个IVa bHLH亚支基因大豆和M. Truncatula.[21那28].该发现表明,在皂苷生物合成的演变期间,FABACEAE植物可能已经在皂苷生物合成的演变期间获得了大量亚氏素IVA成员。
本研究对fabids中IVa bHLHs亚支进行了广泛的探索,发现fabids科植物中有大量的IVa亚支成员,系统发育分析将其分为3个类群。组1的成员最多,包括MtTSARs和GubHLH3。组2和组3成员较少,均未进行功能鉴定,但与组1明显不同,在Fabaceae植物中高度保守。我们还进行了硅分析,以阐明其结构和功能。本研究将有助于缩小豆科植物中尚未鉴定的皂苷生物合成调控因子的候选范围,阐明豆科植物IVa亚支系成员的进化。
结果
豆科植物中大量的IVa亚支成员
共鉴定出319个bHLH蛋白和33个IVa亚分支成员g·马克斯[28].我们得到355个序列g·马克斯bHLH蛋白(附加文件1:表S1)使用PlantTFDB [31].然后,我们根据全长蛋白的序列相似性为新颖成员分配给新型成员并重新选择的子项态IVA成员。虽然五种蛋白质(GmbHlH60-64)被指定为前一项研究中的亚洲IVA成员[28],其氨基酸序列相对较长(588-653 aa),与bHLH蛋白聚类更紧密A.拟南芥在系统发育树上(附加文件3.:图S1)。GmbHLH327、329、331、334、337和345是基于BLAST搜索结果新分配给IVa子分类的。最后,我们确定了34g·马克斯亚洲IVA BHLHs从195到390 AA的肽长度范围(附加文件1:表S1)。
我们收集了40种植物的所有bHLH蛋白序列A.拟南芥那C. Roseus.那C.藜麦和各种fabids(附加文件1:表S2)。这些蛋白质被用来查询BLAST搜索中发现的4个和34个亚基IVa bHLHsA.拟南芥和g·马克斯,我们从而识别每种植物物种中的亚洲IVA成员(附加文件2). 豆科植物有61-355个bHLHs和4-35个亚纲IVa成员,而豆科以外的物种有94-250个bHLHs和2-8个亚纲IVa成员(表1)1).因为基因组序列落花生hypogaea和豇豆属unguiculata在PlantTFDB中没有用于预测,他们的bHLH序列可能没有全部收集到。在Fabaceae和非fabids中,IVa亚支基因相对于所有bHLH基因的比例分别为5.56 ~ 18.2%和1.82 ~ 5.76%1).Fabaceae的基因组中IVa bHLH亚支基因的含量明显高于近缘植物科(Mann-Whitney U检验、你 = 329,P.< 10− 9).
在Fabaceae植物中发现了三组亚亚伊氏菌属BHLH
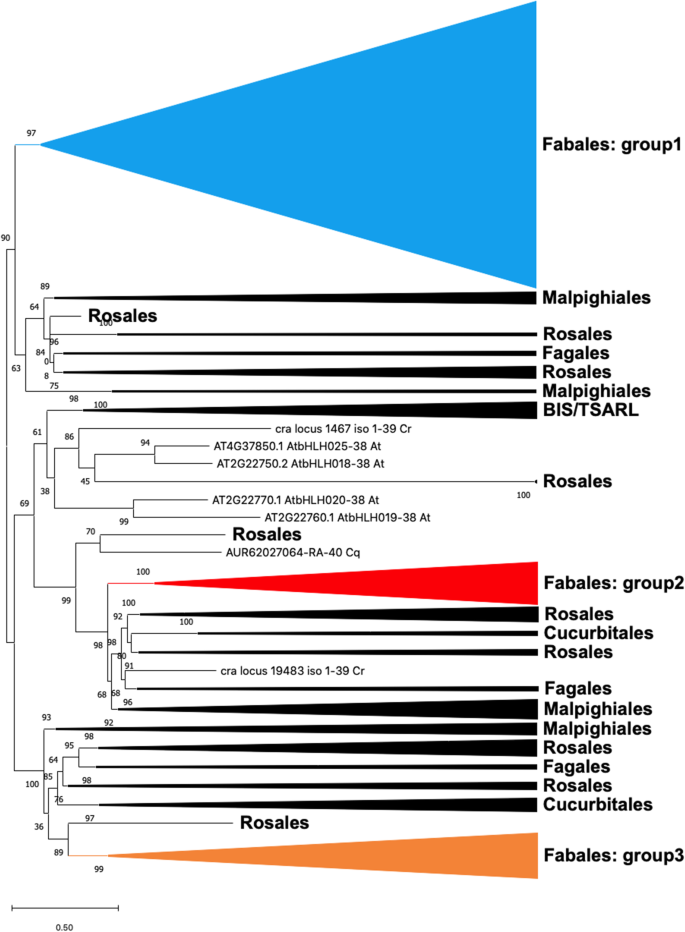
为了观察fabidae和其他fabids中IVa亚支成员的多样性,我们利用全长序列构建了系统发育树。1额外的文件3.:图S2)。次级IVa bHLHs进一步分为三组。大多数Fabaceae亚支IVa bHLHs被纳入第1组(表1),包含所有MTTSAR和GUBHLH3。2组和3人数有限,成员数量有限,但在Fabaceae植物中受到高度保守(附加档案3.:图S2)。
fabids IVa亚支bHLH成员的系统发育树。使用FastTree建立近似极大似可能树,并使用MEGA x进行可视化,使用1000个重样本和Shimodaira-Hasegawa检验计算每个节点的局部支持值。详情见附加文件3.S2:无花果。
bHLH和ACT-like结构域和外显子-内含子结构的守恒
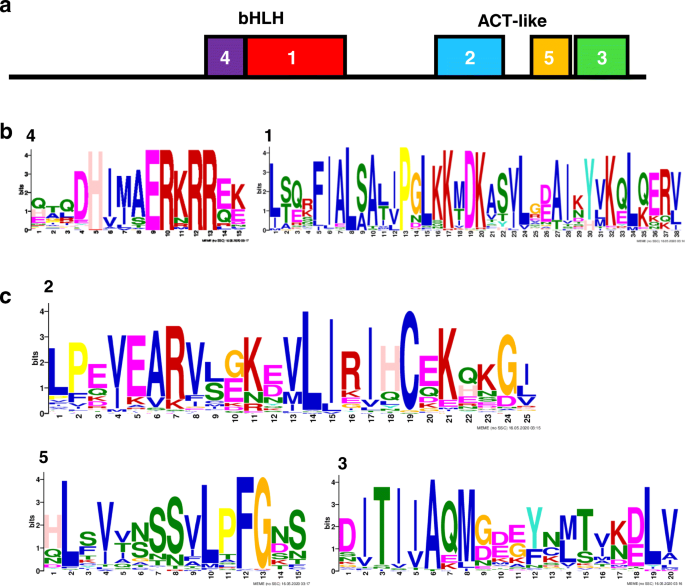
如先前研究所述[16那28],BHLHS具有高度保守的蛋白质结构域与同一亚洲的其他成员。Subclade IVA BHLH包含BHLH域和C终端“样式域;基本区域联系人独联体- 基因组DNA上的微生物,而HLH和作用样域涉及二聚化[18那25那32那33].使用MEME算法[34[我们搜索了这些保守的域名(图。2额外的文件3.:图。S3)在82个亚洲IVA BHLH中g·马克斯那M. Truncatula.,l .对虾(附加文件1:表S1)。我们发现在几乎所有82种蛋白质中都有5个非常保守的基序(图。2a) ;基区和HLH区的两个上游基序(图。2b), c端对应ACT-like域的三个基序(图5)。2c).一些组1成员,GmbHLH105和106和LjbHLH021,缺少基本区域(附加文件3.:图。S3)和这些三种蛋白在系统发育树中聚集在一起(附加文件3.:图S2)。
bHLH和ACT-like域。使用MEME进行领域预测和可视化。(a) IVa bHLHs亚分支结构简化。(b) bHLH结构域和(c) ACT-like结构域中的保守氨基酸残基。详情见附加文件3.S3:无花果。
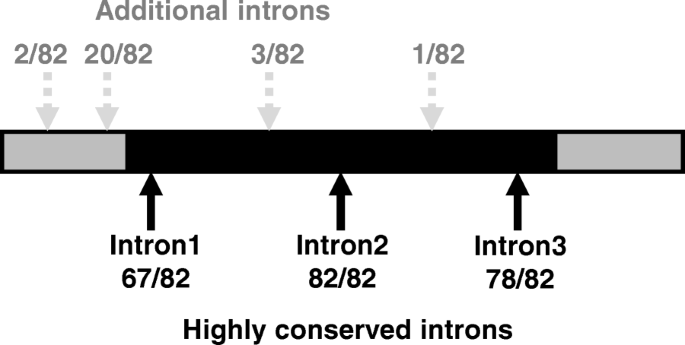
我们证实了IVa亚型bHLH基因的外显子/内含子结构是保守的,但也有一些例外。3.).大多数成员有4个外显子和3个内含子。所有82个IVa bHLH亚支基因均在HLH结构域内包含1个内含子,但其长度高度可变(附加文件1:表S3)。这个保守的内含子位置对应于模式D,如之前的一项研究所定义的[28].MtbHLH138型那MtbHLH177那gmbhlh334,LjbHLH014缺少内含子3和外显子4(附加文件1:表S3),导致不完整或缺少类似act的域(附加文件3.:图S3)。由于组1、组2和组3的一些成员获得或缺少内含子(附加文件1:表S3),结构多样化可能是在演化过程中独立发生的。
简化exon-intron组织。编码序列(CDSs)和非翻译区域(utr)分别用黑色和灰色盒子表示。箭头表示82个IVa亚支成员中保存良好的内含子的位置g·马克斯那M. Truncatula.,l .对虾.灰色虚线箭头显示在某些亚维成员中发现的内含子的位置。详情见附加文件1:表S3
基于跨组的高度保守的蛋白质领域和外显子组织,我们确认第1,2和3组无疑是亚洲IVA的成员。
各组bHLH基因表达模式
使用公开可用的表达图谱g·马克斯那M. Truncatula.,l .对虾,我们比较了每种植物中同源基因的表达模式(表1)2).组1的同源基因在不同植物种间没有完全保守的表达谱。例如,尽管Tsar1.(MtbHLH150型)在叶片和叶柄中表达量较高,其同源基因、LjbHLH054型和Gmbhlh345,分别在结节和花中最高(附加文件3.:图S4)。组2成员通常在结节中表达,除了gmbhlh116和gmbhlh334,未观察到表达。第3组几乎所有基因都在地下组织即根和根瘤中表达。基因组3的四个基因中的三个g·马克斯也在豆荚中表达。
讨论
bHLHs是植物转录因子家族中最具多样性的一个,它调节着生物过程的许多方面,包括器官发育、特殊代谢和对环境刺激的反应[19].次级分支IVa bHLH成员似乎调节专门的新陈代谢和防御反应[5.那19]. 在这项研究中,我们发现豆科植物在其基因组中拥有比其他豆科植物更多的IVa bHLH亚类基因(表1)1,无花果。1).g·马克斯和甘氨酸Soja.与其他Fabaceae相比,bHLHs和IVa亚支成员的数量大约增加了一倍,因为他们经历了两次全基因组复制事件,基因组大小增加了一倍[35那36].虽然bhh的数量在M. Truncatula.与其他豆科植物相似,在Medicago基因组(表1).因此,M. Truncatula.可能在溶血皂苷生物合成途径的发展过程中,从大豆皂苷途径复制了其次级IVa bHLHs(附加文件3.:图。S5)。
在82个IVa亚支成员中,结构域结构和外显子-内含子组织高度保守g·马克斯那M. Truncatula.,l .对虾(无花果。2那3.).Fabaceae Subclade IVA BHLH蛋白在系统发育树中清楚地分为三组(图。1).我们发现属于组1的Fabaceae bHLHs的数量有很强的偏倚,尽管在其他fabids中没有发现这种偏倚(表1).组1可能是一组调控豆科植物皂苷生物合成的转录因子,因为所有的MtTSARs和GubHLH3都包含在这组中(补充文件3.:图S2)。此外,第1组同源基因的表达模式也不保守(Table . 1)2),而大豆皂苷生物合成调控因子GubHLH3不是与MtTSAR1最接近的同源物[22]. 因此,尽管组1成员的重复明显发生在祖先的豆科植物中,但它们的表达模式和对皂苷生物合成的贡献可能在物种形成后发生了分化。因此,我们应该在第1组成员中寻找候选的大豆皂甙生物合成调节剂。
第2组和第3组的成员较少,但高度保守(图。1, 桌子1)并且倾向于在根瘤和根中表达(表1)2).我们确认了合作表达LjCYP93E1(豆属asaponin生物合成基因)和LjbHLH032(组2亚分支IVa bHLH),皮尔逊相关系数为0.797(附加文件3.:图S6)。此外,豆科植物三萜皂苷可能在根际发挥重要作用,已有研究报道;皂苷积累增加,结节增多[16豆属asaponins是根渗出物的主要成分[37].这些观察表明,第2组成员通过调节大豆皂苷的生产来影响根际的生物相互作用。一般而言,bHLH蛋白形成同源二聚体和异源二聚体,调控靶基因的表达[18那25那32那33].2组和3组的IVa亚支系成员也可能调控豆科植物皂苷的生物合成,值得进一步研究。
Fabaceae拥有更多的亚维成员,尽管Fabaceae和非Fabaceae之间的BHLH基因总数没有显着差异(Mann-Whitney U测试,你 = 210,P.= 0.1639)。这说明豆科其他亚支的基因可能较少。根据bHLH结构域的系统发育关系,我们粗略估计了所选物种中每个亚分支中存在的基因数量,发现在任何亚分支中都没有特异性的收缩(补充文件1:表S4)。
结论
在本研究中,我们构建了40种植物的全长IVa bHLH亚支蛋白的系统发育树,主要由fabids组成。结果表明,IVa bHLHs亚支系可分为3个类群,Fabaceae植物中含有大量的第1类群成员,包括目前发现的所有皂苷生物合成调节剂。这些信息将有助于揭示尚未确定的大豆皂苷生物合成调控因子。另一方面,Fabaceae中还没有第2或第3组基因的功能特征。这些基因是阐明Fabaceae亚支IVa bHLH转录因子的进化和功能的重要靶点。
方法
序列检索
具有代表性的蛋白质序列G. Uralensis.是从G. Uralensis.基因组数据库[38].基于从Pfam 32.0下载的HLH结构域(PF00010)的隐马尔科夫模型(HMMs)共检索到163个假定的bHLH蛋白[39那40],使用hmmer v3.3软件[41那42].从PlantTFDB v5.0中检索到其他植物的bHLH结构域序列和bHLH蛋白全长序列(仅为初级亚型)[31那43].利用BLAST搜索对所有的IVa亚分支蛋白进行鉴定A.拟南芥和g·马克斯e值阈值<1e-50。选择的bHLH蛋白列在附加文件中2.
系统发育树分析
使用Clustal Omega v1.2.3对全长bHLHs或bHLH结构域进行蛋白比对[44使用默认设置。使用FastTree v2.1.10生成一个Newick文件[45使用默认设置。系统发育树使用MEGA X从Newick文件中可视化[46].
保守基序和外显子-内含子结构的鉴定
次级IVa bHLHs的保守基序来自g·马克斯那l .对虾,M. Truncatula.使用MEME V5.1.1预测34那47].从植物血红素V12.1检索外显子系统结构[48那49豆类信息系统[50那51].
表达模式分析
从中检索了BHLH基因的表达模式莲花基地(52那53,大豆eFP浏览器[54],Medicago EFP浏览器[55),Medicago Truncatula.基因表达图谱[56那57].
数据和材料的可用性
本研究中分析的所有数据可在方法部分引用的出版物和网站上获得。
缩写
- A.拟南芥:
-
拟南芥
- bHLH:
-
基本螺旋环螺旋
- 爆炸:
-
基本局部对齐搜索工具
- 国际清算银行:
-
BHLH IRIDOID合成
- C. Roseus.:
-
Catharanthus roseus也叫
- C.藜麦:
-
藜藜麦
- 光盘:
-
编码序列
- g·马克斯:
-
大豆
- G. Soja.:
-
甘氨酸Soja.
- G. Uralensis.:
-
乌拉尔甘草
- 嗯:
-
隐马尔可夫模型
- l .对虾:
-
Lotus对虾
- M. Truncatula.:
-
Medicago Truncatula.
- 惩罚:
-
茉莉酸甲酯
- 米娅:
-
Monoterpenoid吲哚生物碱
- 沙皇:
-
三萜皂苷生物合成激活调节剂
- Tsarl:
-
TSAR-like
- UTR:
-
非翻译区
参考
- 1.
Thimmappa R、Geisler K、Louveau T、O'Maille P、Osbourn A。植物中三萜的生物合成。植物生物学年鉴。2014;65:225–57.
- 2.
p450和UGTs:三萜皂苷结构多样性的关键角色。植物生理学杂志。2015;56(8):1463-71。
- 3.
Singh AK, Kumar SR, Dwivedi V等。WRKY转录因子夜蛾通过调节植物甾醇和防御途径来调节甘蔗酰基含量和生物应激耐受性。新植物。2017; 215(3):1115-31。
- 4.
徐杰,吴S,徐y等。BCBZIP134的过度表达负调节索己素蛋白的生物合成。植物细胞组织器官邪教。2019; 137:297-308。
- 5.
bHLH转录因子在茉莉酸信号转导中的作用和功能。实验机器人。2017;68(6):1333-47。
- 6.
González-Coloma A, López-Balboa C, Santana O, Reina M, Fraga BM。Triterpene-based植物防御。Phytochem启2011;10(2):245 - 60。
- 7.
三萜皂苷和皂苷元的结构和溶血活性关系。中华医学杂志。2017;71(1):50-8。
- 8.
Vo NNQ,野村证券,Muranaka T,福岛首席执行官。五环三萜作为环氧化酶和脂氧化酶抑制剂的构效关系。J Nat Prod. 2019;82(12): 3311-20。
- 9.
奥斯巴恩AE,Clarke Br,Lunness P,Scott Pr,Daniels MJ。缺乏百叶香素的燕麦物种易受感染的影响Gaeumannomyces茎var. tritici。植物病理学杂志。1994;45(6):457-67。
- 10.
Shinoda T, Nagao T, Nakayama M等。十字花科植物中三萜皂苷的鉴定Barbarea寻常魅力,作为对小菜蛾的摄食威慑,Plutella Xylostella..化学生态学报。2002;28(3):587-99。
- 11.
Huhman DV、Berhow MA、Sumner LW。植物地上和地下组织中皂苷的含量测定Medicago Truncatula..农业化学学报。2005;53(6):194 - 194。
- 12.
MA Berhow, Kong SB, Vermillion KE, Duval SM。大豆中a、B组大豆皂苷完全定量。农业化学学报。2006;54(6):2035-44。
- 13.
CYP716A179是一种三萜C-28氧化酶,在植物组织培养的匍匐茎中起重要作用乌拉尔甘草.植物细胞批准。2017; 36(3):437-45。
- 14
铃木H,福岛行政长官,清水Y,等。Lotus对虾三萜的结构和表征CYP716A51和LjCYP93E1参与植物生物合成的基因。植物生理学杂志。2019;60(11):2496-509。
- 15
Seki H, Sawai S, Ohyama K,等。甘草三萜功能基因组学鉴定参与甘草酸生物合成的CYP72A154植物细胞。2011;23(11):4112 - 23所示。
- 16
conalonieri M, Cammareri M, Biazzi E等。转基因木桶植物中三萜皂苷的生物合成和根瘤的发生Medicago Truncatula.Gaertn.)表达一种新的β -amyrin合酶(阿索沙1基因。植物Biotechnol J. 2009; 7(2):172-82。
- 17
Delis C,Krokida A,Georgiou S,等。羽扇豆醇合成酶在糖尿病中的作用Lotus对虾结节的形成。新植醇。2011;189(1):335 - 46所示。
- 18.
Heim MA,Jakoby M,Werber M,Martin C,Weisshaar B,Bailey PC.植物中的基本螺旋-环-螺旋转录因子家族:蛋白质结构和功能多样性的全基因组研究。分子生物学。2003;20(5):735–47.
- 19.
植物中碱性螺旋-环-螺旋蛋白的起源和多样性。中国生物医学工程学报。2010;27(4):862-74。
- 20.
Mertens J, Pollier J, Vanden Bossche R, Lopez-Vidriero I, Franco-Zorrilla JM, Goossens A. bHLH转录因子TSAR1和TSAR2调控三萜皂苷的生物合成Medicago Truncatula..植物杂志。2016;170(1):194 - 210。
- 21.
Ribeiro B, Lacchini E, Bicalho K等。三萜皂苷生物合成的种子特异性调节因子Medicago Truncatula..植物细胞。2020;32(6):2020 - 42。
- 22.
田村,吉田,平冈等。碱性螺旋-环-螺旋转录因子GubHLH3正向调控大豆皂苷生物合成基因乌拉尔甘草.植物生理学杂志。2018;59(4):783-96。
- 23.
Jarvis DE, Ho YS, Lightfoot DJ等。的基因组藜藜麦.大自然。2017;542(7641):307 - 12所示。
- 24.
Van Moerkercke A,Steensma P,Schweizer F,等。BHLH转录因子BIS1控制单调吲哚生物碱途径的虹膜骨分支Catharanthus roseus也叫.美国国家科学院学报2015;
- 25.
Van Moerkercke A, Steensma P, Gariboldi I等。基本的螺旋-环-螺旋转录因子BIS2是药用植物中单萜吲哚生物碱生产所必需的Catharanthus roseus也叫.植物j . 2016; 88(1): 3 - 12。
- 26
Mertens J, Van Moerkercke A, Vanden Bossche R, Pollier J, Goossens A. Clade IVa基本的螺旋-环-螺旋转录因子组成了一个保守的茉莉酸信号通路的一部分,用于调节具有生物活性的植物萜类生物合成。植物生理学杂志。2016;57(12):2564-75。
- 27
Fiallos-Jurado J, Pollier J, Moses T,等。中药材中皂素合成基因的含量测定、表达分析及功能鉴定藜藜麦树叶。植物SCI。2016; 250:188-97。
- 28
哈德逊卡,哈德逊我。大豆基本螺旋-环-螺旋转录因子的分类。国际基因组学杂志。2015;
- 29
孙辉,范宏杰,凌汉平。番茄bHLH基因家族的全基因组鉴定与特性分析。BMC基因组学。2015;16(1):9。
- 30.
张超,冯锐,马锐,等。桃基本螺旋-环-螺旋超家族成员的全基因组分析。《公共科学图书馆•综合》。2018;13 (4):e0195974。
- 31.
植物转录因子数据库。http://planttfdb.cbi.pku.edu.cn/. 查阅日期:2020年5月8日。
- 32.
Feller A, Hernandez JM, Grotewold E. An ACT-like结构域参与了几种植物碱性-螺旋-环-螺旋转录因子的二聚。中国生物医学工程学报,2006;
- 33.
Kong Q, Pattanaik S, Feller A,等。关键词:玉米转录因子,基本螺旋-环-螺旋,act结构域,二聚体,调控开关
- 34.
MEME套件。http://meme-suite.org/index.html.. 查阅日期:2020年5月16日。
- 35.
Schmutz J, Cannon SB, Schlueter J,等。古多倍体大豆基因组序列。自然。2010;463(7278):178 - 83。
- 36.
谢敏,Chung CY,李明伟,等。参考级野生大豆基因组。Nat Commun。2019;10(1):1216。
- 37.
Tsuno Y,Fujimatsu T,Endo K,Sugiyama A,Yazaki K. Soyasaponins:在大豆中的一类新的根渗出物(大豆).植物生理学杂志。2018;59(2):366-75。
- 38
Mochida K,Sakurai T,Seki H,等。基因装配和注释草案乌拉尔甘草,一种药用豆科植物。植物J。2017;89(2):181–94.
- 39
El Gebali S,Mistry J,Bateman A,等人,《2019年Pfam蛋白质家族数据库》,核酸研究,2019年;47(D1):D427–32。
- 40.
包含了。https://pfam.xfam.org/.2020年5月6日通过。
- 41.
HMMER web服务器:交互式序列相似性搜索。核酸研究2011;39(Web服务器版):W29-37。
- 42.
HMMER。http://hmmer.org/.2020年5月6日通过。
- 43.
田F,杨立德,孟YQ,金吉,高G. PlantRegmap:植物中的职能监管地图。核酸RES。2020; 48(D1):D1104-13。
- 44.
Sievers F, Wilm A, Dineen D等。使用Clustal omega快速,可伸缩的高质量蛋白质多序列比对一代。Mol Syst Biol, 2011;7:539。
- 45.
价格Mn,Dehal PS,Arkin AP。FastTree 2 - 大致校准的最大似然树木。Plos一个。2010; 5(3):E9490。
- 46.
Kumar S,SteCher G,Li M,Knyaz C,Tamura K. Mega X:计算平台的分子进化遗传分析。mol Biol Evol。2018; 35(6):1547-9。
- 47.
Bailey TL,Elkan C.通过期望最大化拟合混合物模型,以发现生物聚合物中的图案。Proc Int Conv Intel Syst Mol Biol。1994年; 2:28-36。
- 48.
等。植物基因组:绿色植物基因组学的比较平台。核酸研究2012;40(数据库版本):D1178-86。
- 49.
Phytozome v12.1。https://phytozome.jgi.doe.gov/pz/portal.html.访问于2020年5月18日。
- 50.
Dash S,Campbell JD,Cannon Ek等。豆类信息系统(legumeinfo.org):豆类家族的一组联合数据资源的关键组成部分。核酸RES。2016; 44(D1):D1181-8。
- 51.
豆科植物信息系统。https://legumeinfo.org/.访问于2020年5月18日。
- 52.
关键词:波特曼;古普塔;莲花基地:豆科模型的集成信息门户Lotus对虾.Sci众议员2016;6:39447。
- 53.
莲花根据。https://lotus.au.dk/.2020年5月25日通过。
- 54.
大豆eFP浏览器。http://bar.utorto.ca/efpsoybean/cgi-bin/efpweb.cgi..访问19月19日19日。
- 55.
Medicago eFP浏览器。http://bar.utoronto.ca/efpmedicago/cgi-bin/efpWeb.cgi.访问19月19日19日。
- 56.
何杰、本尼迪托·瓦、王明等Medicago Truncatula.基因表达图谱网络服务器。生物信息学。2009;10:441.
- 57.
Medicago Truncatula.基因表达图谱。https://mtgea.noble.org/v3/.访问20月20日20日。
致谢
不适用。
资金
国家自然科学基金(no . JP19J10245)、国家自然科学基金(no . JP20H02913)和国家自然科学基金(no . JP19H02921)资助。
作者信息
从属关系
贡献
HSe和TM参与并监督研究;HSu、HSe和TM设计了研究;徐进行了分析;徐克写了手稿;HSe和TM进行了手稿修订。所有作者都阅读并批准了最后的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者声明他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
表S1。
编号g·马克斯那M. Truncatula.,l .对虾BHLH基因。表S2。亚洲IVA BHLHS亚核发育树分析的物种列表。表S3。Exon-intron组织。CDSs中含有额外内含子的基因用红色表示。括号中给出了这些额外内含子的长度。HLH域内的内含子用黄色突出显示。表S4。每个亚克士中的基因数量。
附加文件2补充数据S1。
362个IVa bHLHs亚分支的蛋白序列用于系统发育树分析。
附加文件3图S1。
bHLH蛋白iif和IVa亚支系统发育树大豆和拟南芥.图S2。亚洲亚洲亚氏素母植物细节素描树在法庭花。图S3。用模因鉴定IVa-bHLH亚类蛋白质的预测结构域。图S4。TSAR1同源物的表达模式。数据是从莲花基础,大豆eFP浏览器,和Medicago eFP浏览器。图S5。豆类皂苷和溶血性皂苷的糖易系统的生物合成途径M. Truncatula..该图显示了豆属asaponins和溶血性皂苷的代表性糖蜜。已经发现细胞色素P450单氧基酶用于氧化β-氨基骨骨干的不同碳位置[细胞色素P450酶(待氧化的位置),*在大豆中表征]。虽然豆科皂苷途径仅是泥质植物,但仅MedicagoSPP。获得溶血途径。图S6。的表达LjCYP93E1和LjbHLH032.数据检索莲花根据。Lj1g3v3555800:LjCYP93E1;lj0g3v0292969:LjbHLH032。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Suzuki,H.,Seki,H.&Muranaka,T.洞察Fabaceae亚洲IVA BHLH转录因子的多样化。BMC植物生物学21,109(2021)。https://doi.org/10.1186/s12870-021-02887-021-02887-021-02887-021-0287-0.
关键字
- 蝶形花科
- 三萜皂苷
- Bhlh.
- 转录调节
- 分类