抽象的
背景
小麦收获前发芽对产量和品质都有严重的危害。对小灵通的抗性是由分布在所有21条小麦染色体上的许多基因控制的数量性状。本研究针对一个大效数量性状位点(QTL)QPHS.CCSU-3A.1.使用先前发育近代源线(NIL)的几套的pHS抗性。通过RNA测序对授粉后15,25和35天的种子进行转录组分布的RNA测序检查两对含有ISOLINE的缺点,以鉴定QTL下面的候选基因,并阐明对pHS抗性的基因作用.在每个DAP,研究了分离丝素之间的差异表达基因(DEGS)。
结果
基因本体和Kegg途径浓缩关键Degs的分析表明,六个候选基因基础QPHS.CCSU-3A.1.负责小麦的pHS抗性。通过定量RT-PCR进一步验证候选基因表达。在靶向QTL间隔内,16种遗传变体,包括五种单一核苷酸多态性(SNP)和11个吲哚,在抗性和易感中的测量中显示出一致的多态性。
结论
该QTL中含有与激素信号通路相关的核心基因,可作为分子标记辅助选择的关键基因组区域。本研究检测到的候选基因和SNP/indel标记为了解小麦小灵通抗性机制和标记辅助育种提供了宝贵资源。
背景
小麦(Triticum aestivum.是世界上主要的谷类作物。收获前发芽(PHS)会严重影响产量及其营养和加工质量,在全世界每年造成超过10亿美元的损失[1,2].因此,pHS抗性是小麦遗传研究和育种的重要特征[3.,4].
种子休眠和萌发,有关pHS的两个主要方法,受到许多环境和分子因素的调节;其中,内源性激素平衡,特别是在脱落酸(ABA)和赤霉素(GA)之间起到至关重要的作用[5,6].在谷类中,ABA参与成熟种子的休眠发育和水解酶合成抑制[7],而GA促进种子储备的新陈代谢,并诱导种子萌发的水解酶合成[8].除了植物激素转导基因外,许多转录因子(TFs)也参与小灵通的调控,如由aba不敏感(ABI)基因编码的B3结构域、AP2结构域和bZIP因子类Abi3.,Abi4.,Abi5., 分别 [9,10.] TFIIS转录伸长因子II编码减少休眠2.(RDO2.)[11.和光敏色素相互作用因子(PIFs) [12.].
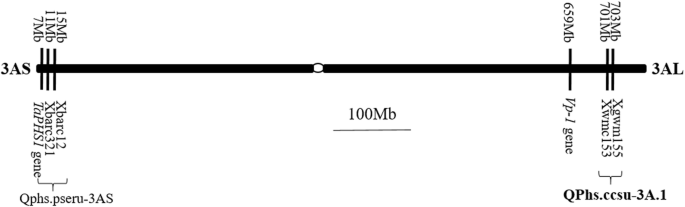
对小灵通的抗性由数量性状位点(QTL)控制[13.,14.,15.位于面包小麦所有21条染色体上的染色体;其中,第3和第4染色体组的QTL一致解释了较大的表型变异[16.,17.,18.,19.].在4AL位点上已经鉴定出几个候选基因,其中包括两个种子休眠基因PM19-A1和A2通过转录组分析[20.和一种原因性的种子休眠基因MKK3.位于旁边PM19通过比较基因组学方法[21.].Wang等人。[22.使用基因分型和表型表征在近似同学线(NIL)的多对的基因分型和表型表征的主要4BL QTL鉴定了五个候选基因。对于第3组染色体,3AS的主要轨迹,解释了23-38%的表型变异,使用红粒父母的交叉源性群群体鉴定了表型变异[23.].后来,刘等人。[24.]克隆了一个基因(Taphs1.)来自3as qtlQphs.pseru-3AS.第三组染色体上的其他已知基因包括胎生(VP-1) 要么Abi3.[25.]在染色体的长臂上,它作为小麦晚期胚胎发育的调节器。Kulwal等人。[26.]报道了从pr8198(小灵通抗性)/ HD2329(小灵通敏感)细粒系中分离到的3AL主要小灵通抗性QTL。QPHS.CCSU-3A.1.六个经过六个测试环境中的表型变异的78.03%,并且位于XWMC153和XGWM155的标记间隔内从厘米的致纳米〜183厘米的遗传距离[26.].这个位于染色体臂3AL上的主要QTL尚未克隆和鉴定。1).
在我们以前的一项研究中,我们开发了几套抗QTL的抗性和敏感的零QPHS.CCSU-3A.1.[27.].同基因的线(NILs)对线附近具有相同遗传背景除了目标轨迹,和尼尔斯·对比特征性能可以定量的特点孟德尔因素,这使得他们理想的遗传资源识别候选基因和标记潜在目标QTL(密切相关22.,27.].RNA测序(RNA-SEQ)是检测差异表达基因(DEGS)和新型表达基因的强大方法,并且广泛用于转录组研究[28.,29.,30.].来自转录组分析的所有基因的表达趋势将是对基因功能的深入研究和它们在复杂生物过程中的相互作用网络的有价值的数据[31.].对比度基因型的转录组分析可以揭示用于分子反应的相关信号途径,导致应力下的生物化学和形态学变化[29.,30.].此外,NILs上的RNA-seq可以准确检测QTL区域内的DEGs和QTL连锁的单核苷酸多态性(SNPs),因此它已被用于识别许多作物的候选基因和标记[20.,32.].
在这项研究中,我们使用了两对含有的含有的NIL,其中ISOLINE之间的PHS性能具有极显显着差异,以研究其在授粉后15,25和35天的种子上进行转录组谱。父母线Chara和DM5637B * 8用于开发NIL的含有白色品种,这消除了pHS抗性和红色基因之间相关性的可能性。该研究的目的是:1)分析不同种子发育阶段的NIL之间的DEG,以提供对pHS抗性的洞察力,2)通过在不同种子发育阶段的表达分析中验证候选基因,以及3)检测可以区分的SNP和INDELS可以区分QTL间隔内的抗性和易感性分离量用于小麦的标记辅助育种pHS抗性。
结果
转录组合汇编质量和映射统计
在质量控制后,共有36个样品产生304 GB的高质量150-BP配对结束序列读数,平均为每个图书馆的5600万清洁读数。近98和96%的清洁读数分别具有Q20和Q30的质量得分。大约70%的测序读数被映射到小麦参考基因组,其中包括55%,具有独特的匹配。每个图书馆检测到的转录物总数范围为72,485至96,979,占所有小麦基因的近60%。Pearson为每个组合的三个生物重复之间的相关系数范围为0.84至0.99,表明三个重复的一致性。
与pHS抗性有关的差异基因表达
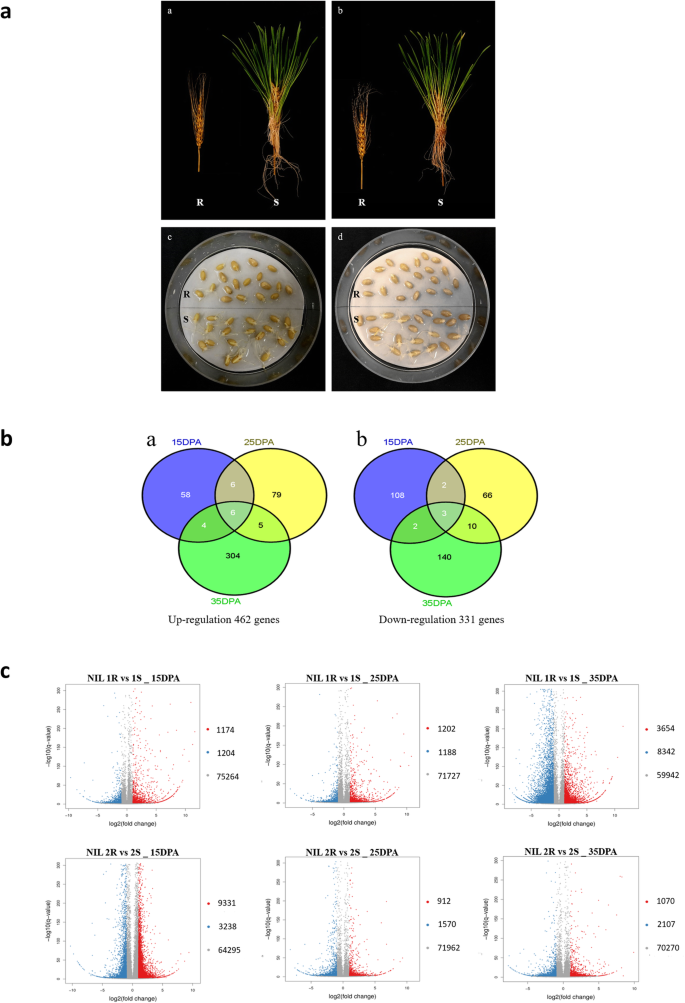
对比等值线中的差异基因表达汇总见表1和图。2.在15dPa,在两个零对中通常检测到抗性('R')和易感('r')和易感('s')中的1195摄氏度。在35dPa下检测分离滤体之间的类似数字(1298)℃。然而,在两对对中,在25dPa中检测到更少的egs(776)。识别QTL下面的基因QPHS.CCSU-3A.1.特别注意,在氮对两对中位于染色体臂3Al上的常见段。在15,25和35dPa分别鉴定了12,12和25个这样的基因。其中,基因traests3a01g462000和traests3a01461400始终上调('R'中的基因表达明显高于'solossine,即'r> s')和基因TRAESK3A01G466700在三个时间点上均持续下调(R′同线基因表达显著低于S′同线基因表达,即‘R
NIL对1和2的比较。A.抗性(R)和易感易感性的表型差异两种NIL对。(a)和(b)分别是在测试的第7天分别的Nil对1(左)(左)和零成对2(右)的尖峰发芽试验;(c)和(d)在试验的第2天进行种子萌发试验。B. venn图,显示差异表达的基因(DEGS)的数量,其通常(a)在抗性中测量的上调和(b)与易感性分子中的耐腐蚀性分离物中的调节。括号内的数字表示在每个时间点表达的基因数。在每个Venn图的底部注意到的总数。C.在不同时间点显示每个NIL对中的DEG的火山图。X轴表示LOG2变换折叠变化。y轴代表-log10转换p价值意义。蓝点代表上调的舞台。红点代表下调的DEG。灰色点代表非舞台。DPA =黄色假期后天
基因表达在每种乳氨碱中的时间点之间显着差异,包括25dPa和15dPa(25/15)之间的次数,在35dPa和25dPa(35/25)之间的次数,并且在35dPa和15dPa之间的次数(35 /15),研究,特别是对于3AL QTL间隔内的那些。展现了一种表现出值得注意的特征的特殊重点,包括与激素转导和PHS调节TFS(如BZIP TFS和B3或AP2域的TFS)相关的那些,与所识别的SNP和吲哚变体相关的那些,以及那些在不同时间点之间显示ISOLINE之间的显着差异。有趣的是,TraesCS3A01G461400在35/15位点的‘R’等值线表达下调,但在35 DPA位点的‘R’等值线表达显著高于两对NIL的‘S’等值线。在35/15的“R”或“S”等值线中,其他基因共享相同的上或下调节TraesCS3A01G459200(在15和25 dpa的'r'isolines中下调'r'solocine,以及'r> s'),TraesCS3A01G470400(在'soloclines中下调,并在15和25 dpa的'r S1).
deg的功能注释
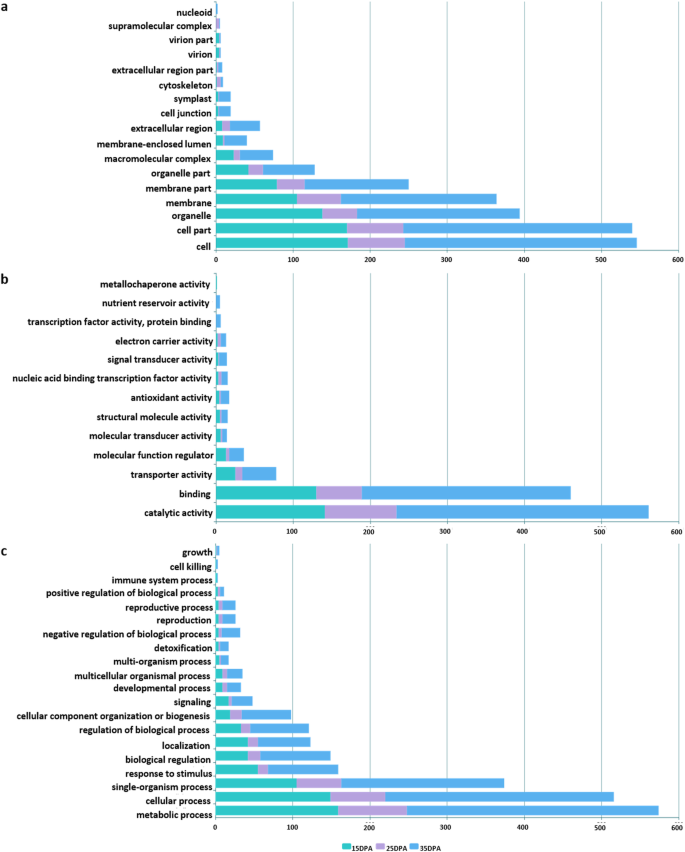
基于GO描述,DEG在功能上分为三个主要类别:细胞分量,分子函数和生物过程(图。3.).
细胞,细胞部分,细胞器,膜和膜部分是细胞组分类别中最常见的术语。催化活性,结合和转运蛋白活性在所有三个时间点都是分子函数类别中最丰富的术语。与生物过程类别中的GO术语相关的大多数基因都是代谢过程,细胞过程和单有机物过程的子类别。值得注意的是,所有三个类别在15和25dPA中的每个类别中的上调和下调基因的所有上调和下调基因相似。然而,在35dPa下,两种NIL对具有比下调基因更高的基因。
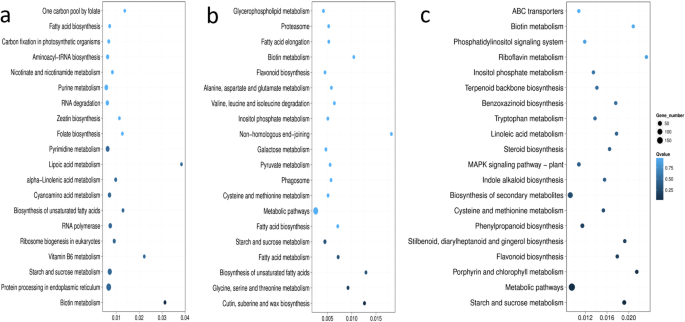
进行途径富集分析以调查分离态之间不同的生物途径(图。4、表2).横跨NIL对的时间点分配给属于五个主要类别的不同途径 - 细胞过程,环境信息处理,遗传信息处理,新陈代谢和有机体系统。其中,新陈代谢是含量中最富集的途径,具有比三个时间点在三个时间点的上调基因更加下调的基因。
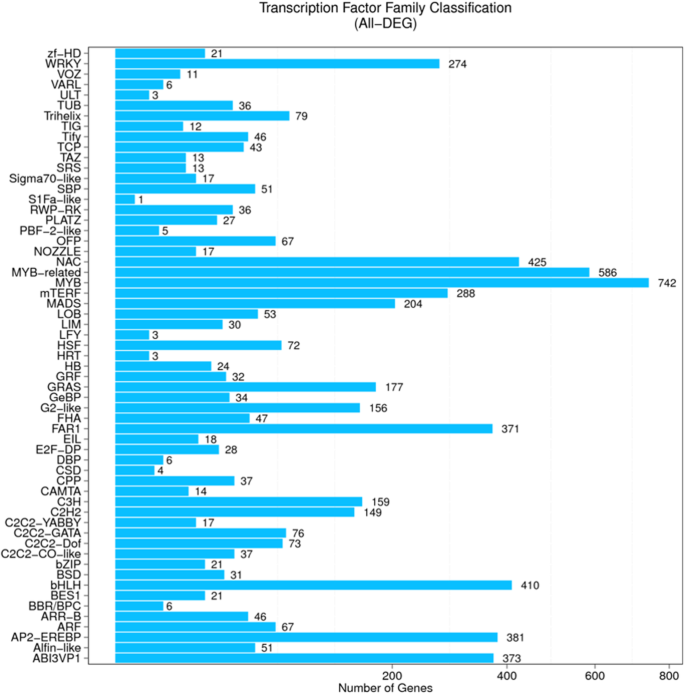
转录因子(TFS)起到控制某些基因表达的分子开关的重要作用,并且在某些环境条件下调节植物生长和发展。在所有ISOLENE中的所有时间点都预测的6050差异表达TFS的广泛数据库搜索所有时间点被分组为59个系列(图。5).MYB和MYB相关转录因子的基因数量最多,分别为742和586个,其次为NAC(425个)和bHLH(410个)。然而,四种广泛表达的转录因子在不同时间点的NIL对中均未表现出一致的DEG模式。
仔细仔细仔细检查在每个DAP中常见的分离物之间的含量;具有与pHS调节途径有关的已知功能的那些,例如植物激素信号转导和MAPK信号传导被认为是潜在的候选基因。基于此,三种其他基因TraesCS3A01G459200,traescs3a01g245000.和traests3a01g225100位于染色体臂3AL上的候选基因(见表)2).
SNP和Indel标记'R'和's中的Isollines之间的多态
检测SNP和吲哚和吲哚显示在两对对中的分子中的分离量之间的可区分基因型。五个SNP和11个衬衫位于目标3AL QTL间隔内或非常接近。其中,在它们的相关基因内发生六种变体(三个SNP和三种诱导),其中五个落入基因外显子,一个在未翻译的区域(UTR)中。虽然其他变体没有重叠在参考基因组中注释的任何基因,但它们向其最近的基因显示出短距离,标记基因距离范围为49至73,788 bp(表3.).
12个基因与SNP和INDELS相关联(表3.).其中8个基因在等值线或时间点之间表现出不同的表达。除了Traescs3a01g449300作为生长素应答因子,其他基因与激素信号通路无关。为TraesCS3A01G449300,在其中任何一个对的'r'和'r'和'sisherines之间没有表达差异(表S1).
qRT-PCR验证候选基因
为了验证RNA-seq的结果,选择6个候选基因进行qRT-PCR检测。相关的表达TraesCS3A01G461400,traests3a01g462000和TRAESK3A01G466700在所有时间点的'r'和's'isolines之间有显着不同,而那个traescs3a01g245000.在25 dpa和35 dpa和35 dpa和35 dpa中显着不同traests3a01g225100在15 DPA处差异显著。值得注意的是,在所有时间点上,的相对表达水平TraesCS3A01G461400分离物之间的两倍不同。所有六个基因显示出一致的表达模式与来自RNA-SEQ分析中获得的那些表达模式(表4).这是在本研究中进行的RNA-SEQ的可靠性的强烈指示。
讨论
候选基因在主要的3AL QTL下面负责pHS抗性
六个候选基因下面QPHS.CCSU-3A.1.本研究根据它们的RNA-seq DEG谱和qRT-PCR验证确定了小灵通抗性。其中,TraesCS3A01G461400在其RNA-seq DEG谱和qRT-PCR表达分析中,' R '和' S '异构体的表达差异都非常显著。Forkhead tf是一个家族,包含一个dna结合域,称为Forkhead box (FOX)。FOX在真核生物中进化保守,是胚胎发育的重要调节因子,受激素信号的影响[33.,34.].与高度保守的FOX结构域相比,forkhead TF蛋白在其序列的其他部分高度分化[35.].在人类中,Forkhead TFS调制信号通路[36.]并且可以是荷尔蒙药物的直接靶向,例如孕激素,以抑制上皮细胞生长[37.].在昆虫中,Forkhead TFS调节激素介导的信号传导,影响碳水化合物,氨基酸和脂肪酸代谢,以及磷脂酰肌醇3-激酶/蛋白激酶B信号通路[38.].在植物中,叉头相关域介导了与类受体激酶的相互作用,进而调控了涉及生长和病原体反应的信号通路;两个研究基因,kapp.(编码激酶相关的蛋白磷酸酶,在体细胞胚胎发生受体激酶1的内化中起作用)和ABA1(编码在ABA生物合成途径中起作用的玉米黄质环氧化酶),是15个已鉴定的含有叉头相关结构域的拟南芥基因之一[39.,40].此外,TraesCS3A01G461400参与嘌呤新陈代谢途径,可在ABA新陈代谢激活中发挥作用[41.,42.].
traests3a01g462000编码一个包含B3域的TF;其家族通过调节ABA和GA代谢来控制胚胎发育和种子成熟[43.].B3 TFS被认为是光合作用的特异性[44.].VP-1是在玉米中鉴定的第一植物基因,其编码B3型TF,其是其他谷物种子成熟期间的ABA信号通路的关键组分。VP-1其外科基因与编码种子储存蛋白,晚期胚胎发生丰富的基因的激活有关,以及花青素生物合成酶的抑制,用于储备动员的发芽后基因,例如。α-淀粉酶和蛋白酶[45.].
TraesCS3A01G461400和traests3a01g462000既是TF基因。TFS在植物生长,发展和对环境压力的反应中发挥重要作用[46.].序列特异性转录因子与其调控基因附近的靶位点的相互作用是基因表达调控的核心机制,生物体通过该机制发展并与环境相互作用[47.].本研究发现的两个TF基因均与激素信号通路相关,且在所有时间点上‘R’的表达均显著高于‘S’等值线,提示‘R’等值线中对基因转录的更积极的调控可能是其抗PHS表型的原因。
有趣的是,另外两种候选基因TraesCS3A01G459200和traescs3a01g245000.功能为受体样激酶(RLKs)。RLKs是表面定位的跨膜受体,调节多种信号通路[48.,49.];有些人与forkhead关联域的互动,例如kapp [40,50.].基因TraesCS3A01G459200涉及许多新陈代谢途径,包括脂肪酸生物合成,淀粉和蔗糖代谢,苯丙醇化生物合成,以及二次代谢物的生物合成(表2),其中据报道,亚苯丙烷丙二醇代谢涉及拟南芥中的原发性种子休眠[51.].基因traescs3a01g245000.通过KEGG富集分析直接参与植物激素信号转导途径。富亮氨酸重复序列(LRR) RLK,由traescs3a01g459200,在拟南芥ABA信号转导中起重要作用;它被ABA上调,其功能丧失导致种子萌发时ABA不敏感[52.].在本研究中,LRR RLK在15和25 DPA的' R '等位基因中表达量显著高于' S '等位基因,但在35 DPA的等位基因中无差异,因为该基因在35/15的' R '等位基因中表达量下调(表1)S1),表明‘R’等值线在早期ABA含量较高。
TRAESK3A01G466700编码参与硫胺素生物合成途径的羟乙基噻唑激酶[53.].参与甘氨酸代谢,硫胺素在能量新陈代谢中具有基本作用,并作为种子萌发的能量储备作用[54.].Golda等人。[54.]发现,在发芽期间,谷物和豆科种子均失去了硫胺素储备的重要组成部分。neumann等。[55.]报道,用硫胺素治疗的种子显着提高了豆类的发芽率Phasenius寻常的.在本研究的所有时间点上,该基因在' R '等值线上的表达量均显著低于' S '等值线上的表达量,这可能意味着在' R '等值线上硫胺素的储量较低,不利于种子的萌发。
traests3a01g225100在ABA诱导的基因表达所需的S型阴离子信道活动中的功能[56.].Kegg浓缩将基因分配给植物Mapk信号通路。MAPK模块直接响应ABA,或与MKK3进行交互;例如,MAP3K16是ABA响应(ABR1)的负调节器,MAP3K17 / 18-MKK3-MPK1 / 2/7 / 14患ABA,衰老和患者拟南芥[57.,58.].MKK3含有NTF2结构域,其主要基因结构在进化期间高度保守[59.].Torada等。[21.]报道MKK3.是小麦种子休眠的主要4AL QTL的致病基因。在这项研究中,traests3a01g225100在15dPa的IS中,表明该基因在种子发育早期阶段,该基因主要发挥对ABA的监管作用。
SNP和indel标记可区分' R '和' S '等值线
“r”和'sishs中的16个SNP或Indel变体中的十种在REFV1.0中没有重叠任何带注释的基因。然而,由于基于RNA-SEQ型材鉴定这些变体,因此它们应该在测试的品种/线的转录基因中。这种反应可能是由于:1)参考品种中国弹簧(Cs)和测试品种/线的基因组之间的大结构变化;或2)Cs不含其他品种中存在的某些基因,例如,PPD-B1和VRN-A1等位基因在CS中不存在[60.].
基因Traescs3a01g449300,作为生长素反应因子(ARF)。生长素通过诱导arf介导的ABI3激活ABA信号,招募arf来控制拟南芥种子休眠[61.].植物因涉及从种子休眠到萌发的过渡,促进种子休眠,抑制种子萌发[62.].虽然其基因表达在染色体中没有差异,但目标主要QTL内的位置表明它通过与坐在该特定基因组区域中的其他信号转导基因相互作用来涉及调节途径。
在QTL区间内,还有一个值得注意的基因组区域,从622,277,558到622,901,141 bp,其中存在一个与基本亮氨酸拉链(basic leucine zipper, bZIP) TFs和ABI5s相关的连续基因块。BZIP TFs由aba介导的信号体激活,并结合特定的顺式作用序列,称为脱落酸响应元件(ABREs)或富含gc的偶联元件,从而影响其下游靶基因的表达[63.].ABIS参与ABA信令,其中一些是TFS;ABI5是已经鉴定的六种类别的这种TFS之一,对于ABA或种子特异性基因表达至关重要[64.].ABI5的转录抑制与降低种子对ABA的敏感性有关,从而导致小麦种子从休眠到萌发的转换[65.].在本研究的三个时间点上,这些基因的对比等值线之间没有一致的表达差异(见表)S1).然而,随着核心ABA信号中的基因函数并且出现在靶向QTL间隔内的簇中,它意味着多种基因和基因相互作用可能使QTL负责pHS抗性。
目标QTLQPHS.CCSU-3A.1.,解释高达78.03%的表型变异[26.],可以被利用为标记辅助选择的关键基因座。QTL区域中的许多基因包括所鉴定的候选基因,SNP / Indel标记相关基因和物理聚类基因,参与激素感知和信号转导,这进一步证明了轨迹在调节和控制pHS抗性中的意义。对于未来的研究,等位基因表征可以在已知具有QTL的其他基因型中进行,并且转基因方法可用于候选基因的功能性测试。
结论
靶向QTL主要3AL QTL的ILS转录组分析QPHS.CCSU-3A.1.负责pHS抗性显示与激素信号传导和能量代谢有关的六种候选基因。QTL间隔内的十六个SNP或Indel标记显示在PHS性能中的“R”和'r'和'sishile之间的一致可区分等位基因。靶向QTL被证实为种子休眠和pHS抗性的关键基因组区域,因为它含有许多参与ABA信号传导途径的核心基因,其中一些核心基因在对比度分离物之间表达表达显着差异。本研究中所确定的候选基因和SNP / Indel标记对于了解PHS抗性的机制以及用于小麦特征的标记辅助育种有价值。
方法
植物材料和组织采样
在以前的研究中,我们使用靶向3AL QTL的异构自血交族方法生成一组NIL [27.].通过基因分型和表型进行了植物材料的正式鉴定[27.]由麦子专家的所有作者。在本研究中使用了两对含量(每对源自F2个体的单个种子下降),在ISOLINE之间的pHS性能显着对比对比的pHS性能(图。2).两对NIL对均来自于‘Chara/DM5637B*8’群体,本研究命名为‘NIL对1R & 1S’和‘NIL对2R & 2S’,分别与之前研究中的NIL_PHSR3AL_3R & 3S和NIL_PHSR3AL_6R & 6S相匹配[27.].'R'表示携带抗性等位基因的分离物,并且对于那些有易感等位基因的人来说。父母的“凯拉”的种子是从澳大利亚谷物Genebank,Horsham,Victoria,澳大利亚获得,澳大利亚沉积数量,父母的DM5637B * 8'的种子是从澳大利亚的Chertgrain Pty Ltd.获得的。本研究中使用的尼尔的种子在西澳大利亚大学麦片子系列中保存,uwanilta-n11(nil_phsr3al_3r),uwanilta-n12(nil_phsr3al_3s),uwanilta-n17(nil_phsr3al_6r)和uwanilta-n18(nil_phsr3al_6s)。
这些NILs在西澳大利亚州珀斯的西澳大利亚大学的温室中被种植,每个同线有三个生物复制。植株生长条件和表型方法与Wang等人的描述相同[27.].每次飙升都记录了开花日期。在15dPa和25dPa的五个粒子下,从每次重复中的每种分离物中的每分离物中的35 dPa和35dPa的三个粒子被随机收集,立即在液氮中冷冻并储存在-80℃下进行RNA提取。
RNA提取,图书馆施工和illumina测序
使用RNeasy Plus Plant Mini Kit (Qiagen),按照制造商的说明,在DNase处理下,从36个样本(4个基因型× 3个时间点× 3个重复)中提取总RNA。提取的RNA的收率和纯度由NanoDrop 2000 (Thermo Fisher Scientific Inc.,澳大利亚)进行评估,完整性由1% (w/v)变性凝胶电泳和Agilent 2100 Bioanalyzer (Agilent Technologies Inc.,美国)进行检验。这些合格和定量的RNA样本在中国北京基因组研究所(BGI)进行测序。BGI的cDNA合成、150 bp对端测序和原始数据过滤协议与Mia等人的描述相同[30.].将生成清洁数据作为FASTQ文件,并计算Q20,Q30和GC内容。在这些清洁数据上进行下游分析,可在国家生物技术信息中心(NCBI)网站上提供,SRA登录PRJNA554312(https://www.ncbi.nlm.nih.gov/sra/prjna554312.).
序列数据分析和DEG识别
高质量的读数被映射到面包小麦参考基因组序列,国际小麦基因组序列联盟(IWGSC)Refseq V1.0(https://wheat-urgi.versailles.inra.fr/)[66.,使用HISAT2 v2.0.4 [67.].Bowtie2完成了读取与参考序列的对齐[68.].采用RSEM v1.2.12计算基因表达量[69.]默认参数。根据Wang等人的Degseq鉴定了DEGS。[70],参数如Mia等人所述。[30.].Up- and down-regulations of DEGs between the isolines were based on the comparison of ‘R’ isoline to ‘S’ isoline, i.e., if a gene expression in ‘R’ isoline was higher or lower than that in ‘S’ isoline, it was considered upregulated or downregulated, respectively.
功能注释、基因本体论和路径分析
基因本体(GO)和所选择的含量的功能性富集是使用过度测量测试(PHYPER)进行的;具有假发现率(FDR)≤0.01的那些被认为是显着富集的。Kegg注释与Mia等人所述相同。[30.].要识别来自DEG的TF编码基因,GETORF工具[71.用来找到每个DEG的开放阅读框架(ORF)。然后使用HMMSEarch将ORF与PLNTFDB对齐TF域[72.从所选的基因中鉴定TF编码基因。
SNP和indel标记的发现
为了找到SNP和indel变异,我们将所有的转录本的干净reads都映射到IWGSC RefV1.0的参考基因组序列(https://wheat-urgi.versailles.inra.fr/)使用bowtie2 [68.].SAM工具包用于调用SNP和indel变体。在3A染色体上检测到变异,特别是在Xwmc153和Xgwm155标记区间内的变异。
使用定量RT-PCR(QRT-PCR)分析验证DEGS
选择本研究鉴定的候选基因进行qRT-PCR,验证RNA-seq结果。利用SensiFast cDNA Synthesis Kit (Bioline Australia),按照制造商的方案合成cDNA。使用SensiFAST SYBR试剂盒(Bioline Australia)在ABI 7500 Fast系统上进行qRT-PCR。利用Primer Premier 5.0软件设计基因特异性引物,以小麦肌动蛋白基因作为内源对照进行样本间归一化。在3个时间点,对两对NILs的每条等值线进行3个生物重复。对于qRT-PCR,将所有生物样本的cDNA进行三份比对[73.].在含有10μl2×sensifast SYBR LO-ROX混合物的20μl反应混合物中进行扩增,每个前进和反向引物和100ng cDNA,具有以下循环方案:1个95°C的循环2分钟,95℃的40个循环,5 s和60℃。使用比较CT方法计算相对基因表达[74.].两个样品T检验用于比较不同DPA的'R'和'和''和'SICTINE中的方法之间的表达差异。
数据和材料的可用性
当前研究期间生成和/或分析的数据集可在国家生物技术信息中心(NCBI)网站上获得,SRA登录号为PRJNA554312 (https://www.ncbi.nlm.nih.gov/sra/prjna554312.).植物材料(种子)保存在西澳大利亚大学小麦种子收集。
缩写
- 阿坝:
-
脱盐酸
- 阿布:
-
阿坝不敏感
- DEG:
-
差异表达基因
- FDR:
-
假发现率
- 狐狸:
-
嘴巴盒子
- GA:
-
赤霉酸
- 走:
-
基因本体论
- 零:
-
Near-isogenic线
- ORF:
-
开放阅读框架
- PHS:
-
收获前发芽
- 论坛:
-
Phytochrome互动因子
- QTL:
-
定量特质基因座/基因座
- RNA-SEQ:
-
RNA测序
- SNP:
-
单核苷酸多态性
- TF:
-
转录因子
参考文献
- 1。
黑色MBJ,霍尔默P.预热发芽 - 经济重要性。在:黑色m,编辑器。种子科学,技术和用途的百科全书。牛津郡:CABI出版;2006.528。
- 2。
Biddulph TB,Plummer Ja,Setter TL,Mares DJ。高温和终端水分胁迫对小麦休眠的影响(Triticum aestivum.l .)。中国农业科学(英文版);2012;
- 3.
陈志强,王志强,王志强,等。加拿大硬粒小麦品种收获前发芽抗性的遗传分析。摩尔品种。2014;33:919-29。
- 4.
Chao S,Elias E,Benscher D,Ishikawa G,Huang Y,Saito M,Nakamura T,Xu S,Faris J,Sorrells M.使用重组替代线在四倍体中使用重组取代线的主要效果种子休眠定量性状基因座的遗传映射小麦。农作物科学。2015; 55(1):1-14。
- 5。
foottitt S, Douterelo-Soler I, Clay H, Finch-Savage WE。拟南芥种子的休眠周期受季节性不同的激素信号通路控制。中国科学院院刊。2011;108(50):20236-41。
- 6。
Nonogaki H,Barrero JM,Li C.编辑。种子休眠,发芽和收获前发芽。前植物SCI。2018; 9:1783。
- 7。
Li C, Ni P, Francki M, Hunter A, Zhang Y, Schibeci D, Li H, Tarr A, Wang J, Cakir M, Yu J, Bellgard M, Lance R, Appels R.水稻-小麦-大麦种子休眠和采前发芽的基因比较。功能整合基因组学。2004;4(2):84-93。
- 8。
李志,高艳,张艳,林超,龚东,关艳,胡建军。活性氧和赤霉素对烟草种子萌发的调控作用。植物学报2018;9:1279。
- 9。
Södermanem,Bricard Im,Lynch Tj,Finkelstein RR。拟南芥ABA不敏感4基因在种子和脱落酸响应信号网络中的调节和功能。植物理性。2000; 124(4):1752-65。
- 10。
Duarte Ke,De Souza WR,Santiago TR,Sampaio BL,Ribeiro Ap,Cotta Mg,Da Cunha Badb,Marchaylini PRR,Kobayashi Ak,Molinari HBC。鉴定和表征核心脱胶(ABA)信号分量及其基因表达谱的响应于非生物胁迫Setaria Viridis..SCI REP。2019; 9(1):4028。
- 11.
Liu Y, Geyer R, van Zanten M, Carles A, Li Y, Hörold A, van noker S, Soppe WJJ。通过对拟南芥减少休眠2基因的鉴定,揭示了聚合酶相关因子1复合体在种子休眠中的作用。《公共科学图书馆•综合》。2011;6 (7):e22241。
- 12.
Penfield S,Josse E-M,Halliday KJ。拟南芥原发性种子休眠控制中PIF6的替代剪接变体的作用。植物mol biol。2010; 73(1):89-95。
- 13。
张惠平,张惠平,谢赫SW,沙阿L,马灿(Triticum aestivum.L.)抵抗因子导致收获前发芽的因素。农艺学。2019; 9(3):117。
- 14。
利用种子休眠技术对六倍体小麦群体采前发芽抗性QTL进行优化鉴定。Euphytica。2015;205(1):287 - 309。
- 15.
Rodríguezm,巴勒塞·j,corbineau f,gubler f,Benech-arnold r.谷物中的休眠(不太多,不那么少):关于这个特质背后的机制。种子sci res。2015; 25(02):99-119。
- 16。
安德森J,Sorrells M,Tanksley S. RFLP分析与耐养殖前抗性抗性相关的基因组区域。农作物科学。1993年; 33:453-9。
- 17。
Mares D,MRVA K.绘制与澳大利亚小麦休眠变异相关的定量特质基因座。AUST JAGRIC RES。2001; 52:1257-65。
- 18。
陈超,蔡树华,白国强。小麦地方品种4A染色体上控制种子休眠和收获前发芽抗性的QTL。摩尔品种。2008;21(3):351 - 8。
- 19。
宋永强,石川,孟克武,等。小麦2B染色体采前发芽QTL区间的精细定位。中国科学(d辑:地球科学)2014;
- 20.
Barrero JM, Cavanagh C, Verbyla KL, Tibbits JFG, Verbyla AP, Huang BE, Rosewarne GM, Stephen S, Wang P, Whan A, Rigault P, Hayden MJ, Gubler F.小麦近等基因系转录组分析发现PM19-A1和A2是主要休眠QTL的候选QTL。基因组医学杂志。2015;16(1):93。
- 21。
托拉达A,Koike M,Ogawa T,Takeouchi Y,Tadamura K,Wu J,Matsumoto T,Kawaura K,Ogihara Y.一种种子休眠在小麦染色体4a上的因果基因编码了地图激酶激酶。Curr Biol。2016; 26(6):782-7。
- 22。
王学军,刘红梅,刘国强,刘国强。小麦收获前发芽4BL主要QTL近等基因系的表型和基因型分析。植物学报。2019;19(1):348。
- 23。
Osa M, Kato K, Mori M, Shindo C, Torada A, Miura H.小麦3A染色体上Vp1同源基因的qtl定位。应用计算机学报。2003;106(8):1491-6。
- 24。
Liu S ., Sehgal S ., Li J ., Lin M ., Trick H ., Yu J ., Gill B ., Bai G.一个小麦收获前发芽关键调控因子的克隆和鉴定。遗传学。2013;195(1):263 - 73。
- 25。
Kocheshkova AA,Kroupin Py,Bazhenov Ms,Karlov Gi,Pochtovyy AA,Upelniek VP,Belov VI,Divashuk Mg。小麦 - 小麦草杂交种中的HAVP-1基因的预先收集萌发性和单倍型变异。Plos一个。2017; 12(11):E0188049。
- 26。
Kulwal P,Kumar N,Gaur A,Khurana P,Khurana J,Tyagi A,Balyan H,Gupta P.在面包小麦中染色体3A染色体3A上的收获前萌芽耐受性的主要QTL的映射。Al Appl Genet。2005; 111:1052-9。
- 27。
关键词:小麦,采前发芽抗性,QTL定位,近等基因系植物生态学报。2018;69(9):864-72。
- 28。
LJ,Belfield EJ,江C,棕色C,Mithani A,Harberd NP。RNA测序在六倍体面包小麦中显示的同种型基因表达的图案。BMC基因组学。2014; 15:276。
- 29。
iquebal ma,sharma p,jasrotia rs,jaiswal s,kaur a,saroha m,angadi Ub,sheoran s,singh r,singh gp,rai a,tiwari r,kumar d.rnaseq分析揭示了候选基因的干旱响应分子途径小麦根组织中的调用分子标记。SCI批准。2019; 9(1):13917。
- 30.
MIA MS,刘H,王X,张C,闫G.造影小麦基因型的根转录组谱分析对其自适应策略对水资源赤字进行了见解。SCI代表2020; 10(1):4854。
- 31.
贾米,关吉,翟Z,耿思,张X,毛升,李A。小麦功能基因组学中的下一代测序时代:更新。作物J. 2018; 6(1):7-14。
- 32.
高胜,郑志强,周敏,刘超。大麦1HL抗冠腐病基因的转录组分析。BMC基因组学。2019;20(1):650。
- 33。
Hannenhalli S,Kaestner Kh。福克斯基因的演变及其在发展与疾病中的作用。NAT Rev Genet。2009; 10(4):233-40。
- 34。
戈尔森ml,kaestner kh。Fox转录因子:从发育到疾病。发展。2016; 143(24):4558。
- 35。
Schmitt-ney M. Foxo成为一个家庭的优势:关于功能和进化的考虑因素。在:细胞,Vol。9;2020。
- 36。
Pallauf K,Duckstein N,Hasler M,Klotz L-O,Rimbach G.Flavonoids作为转录因子NRF2,FOXO和PPAR的推定诱导者。oxid med cell longev。2017; 2017年:4397340。
- 37。
Kyo S,Sakaguchi J,Kiyono T,Shimizu Y,Meaida Y,Mizumoto Y,Mori N,Nakamura M,Takakura M,Miyake K,Sakamoto M,Inoue M. Forkhead转录因子Foxo1是孕激素抑制子宫内膜上皮的直接靶标细胞生长。临床癌症Res。2011; 17(3):525-37。
- 38。
尹z-j,东x-l,kang k,chen h,dai x-y,wu g-a,zheng l,yu y,zhai y-f。FOXO转录因子调节激素介导的若虫延迟介导的信号传导。前面的physiol。2018; 9:1654。
- 39。
Shah K,Russinova E,Gadella TW JR,Willemse J,De Vries SC。拟南芥激酶相关蛋白磷酸酶对体细胞胚胎发生受体激酶1.基因开发的内化。2002; 16(13):1707-20。
- 40.
莫里斯·埃尔,乔尔维尔D,Walker JC。Dawdle,一个关联的域基因,调节植物开发的多个方面。植物理性。2006; 141(3):932。
- 41.
嘌呤代谢物尿囊素通过协同激活脱落酸代谢增强非生物胁迫耐受性。植物学报。2014;37(4):1022-36。
- 42.
Takagi H,Ishiga Y,Watanabe S,Konishi T,Egusa M,Akiyoshi N,Matsuura T,Mori Ic,Hirayama T,Kaminaka H等人。含有应激相关的嘌呤代谢物,可以以myC2调节和脱落酸依赖性方式激活茉莉酸酯信号传导。J Exp Bot。2016; 67(8):2519-32。
- 43.
刘X,侯X.在新陈代谢和信号通路中ABA和GA的拮抗调节。前植物SCI。2018; 9:251。
- 44.
植物特异性转录因子的dna结合结构域:结构、功能和进化。植物科学进展。2013;18(5):267-76。
- 45。
Carbonero P,Iglesias-Fernándezr,Vicente-carbajosa J.B3转录因子的AFL亚家族:在AgeniSperm种子中的进化和功能。J Exp Bot。2017; 68(4):871-80。
- 46。
吴军,张震,张强,刘勇,朱兵,曹军,李智,韩磊,贾军,赵刚,孙旭。小麦转录因子FOX水稻品系的产生及耐盐渗透胁迫的系统筛选。《公共科学图书馆•综合》。2015;10 (7):e0132314。
- 47。
Nakagawa S,Gisselbrecht SS,Rogers JM,Hartl DL,Bulyk ML。DNA结合特异性变化转录转录因子的演变。PROC NATL ACAD SCI。2013; 110(30):12349。
- 48。
Goff KE, Ramonell KM。受体激酶在植物防御中的作用与调控。基因调控系统生物学报2007;1:167-75。
- 49。
Greeff C,Roux M,Mundy J,Petersen M.受体样激酶复合物在植物实时免疫。前植物SCI。2012; 3:209。
- 50。
MacGregor Dr,Kendall SL,Florance H,Fedi F,Moore K,Paszkiewicz K,Smirnoff N,Penfield S.种子生产温度调节初级休眠的种子制作温度调节通过种子涂层苯丙醇代谢。新植物。2015; 205(2):642-52。
- 51。
拟南芥丝氨酸-苏氨酸受体激酶与蛋白磷酸酶的相互作用。科学。1994;266(5186):793 - 5。
- 52.
富含亮氨酸重复受体样激酶1 (leucine repeat receptor-like kinase1)是拟南芥脱落酸早期信号转导的关键膜结合调控因子。植物细胞。2005;17(4):1105 - 19所示。
- 53.
Tani Y,Kimura K,Mihara H. 4-甲基-5-羟乙基噻唑激酶的纯化和性质大肠杆菌.Biosci Biotech Bioch。2016; 80(3):514-7。
- 54.
GołdaA,Szyniarowski P,Ostrowska K,Kozik A,Rapała-Kozik M.硫胺素结合和代谢在选定的谷物和豆类的发芽种子。植物理性生物化学。2004; 42(3):187-95。
- 55.
Neumann G,Azaizeh Ha,Marschner H. Thiamine(维生素B1)种子处理增强了豆豆的萌发和幼苗生长(phoudolusulus vulgaris.L.)暴露于浸泡伤害。Zeitschriftfürpflanzenährungundbodenkunde。1996年; 159(5):491-8。
- 56.
Finkelstein RR,Gampala SSL,摇滚CD。种子和幼苗中的脱落酸信号传导。植物细胞。2002; 14(4):S15-45。
- 57。
植物MAPK级联:只是快速信号模块?植物信号学报。2015;10(9):e1062197。
- 58。
崔世文,李淑英,罗妍杰,郑SG,金施。拟南芥MAP3K16和其他盐诱变MAP3Ks对ABA响应具有冗余调控作用。摩尔细胞。2017;40(3):230 - 42。
- 59。
Colcombet J,SözenC,Hirt H. MKK3上多个MAP3K的收敛识别一组新颖的应力MAPK模块。前植物SCI。2016; 7:1941。
- 60.
Díaza,zikhali m,特纳,isaac p,laurie da。影响Photopheriod-B1和vernalization-A1基因的拷贝数变型与小麦的开花时间改变有关(Triticum aestivum.).《公共科学图书馆•综合》。2012;7 (3):e33234。
- 61。
刘X,张h,赵y,风z,李q,杨h-q,栾s,李j,他z-h。通过在拟南芥中诱导ARF介导的ABI3活化来控制种子休眠通过刺激脱离酸信号传导。PROC NATL ACAD SCI。2013; 110(38):15485-90。
- 62。
吴M,吴j,甘Y.一种新的助生功能的新洞察力:从种子休眠到萌发和花卉开口的转变。植物生长调节。2020; 91(2):169-74。
- 63。
Banerjee A,Roychoudhury A.植物中依赖酸依赖性碱性亮氨酸拉链(Bzip)植物非生物胁迫转录因子。原生质。2017; 254(1):3-16。
- 64。
Skubacz A,Daszkowska-Golec A,Szarejko I. ABI5(ABA-Imensitive 5)在植物发育,非生物应激反应和植物雌激素串扰中的作用和调节。前植物SCI。2016; 7:1884。
- 65.
刘A,高F,Kanno Y,Jordan Mc,Kamiya Y,Seo M,Ayele Bt。通过熟练的后羟色剂的对小麦种子休眠的调节由特定的转录开关介导,所述特定转录开关诱导种子激素代谢和信号传导的变化。Plos一个。2013; 8(2):E56570。
- 66.
Apples R,Eversole K,Feuillet C,Keller B,Rogers J,Stein N,Stein N,Choulet F,Distelfeld A,Poland J等。使用完全注释的参考基因组转移小麦研究和育种的限制。科学。2018; 361(6403):661。
- 67.
Kim D,Langmead B,Salzberg SL。Hisat:一种快速拼接对齐器,内存要求低。NAT方法。2015; 12:357。
- 68.
Langmead B, Salzberg SL.快速间隙读对齐与领结2。Nat方法。2012;9(4):357 - 9。
- 69。
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12(1):323。
- 70.
王L,Feng Z,王X,王X,张X.Degseq:用于从RNA-SEQ数据鉴定差异表达基因的R包。生物信息学。2009; 26(1):136-8。
- 71.
EMBOSS:欧洲分子生物学开放软件套件。趋势麝猫。2000;16(6):276 - 7。
- 72.
Mistry J,Finn Rd,Eddy SR,Bateman A,Punta M.在同源性中的挑战:HMMER3和卷绕式线圈区域的收敛演变。核酸RES。2013; 41(12):E121。
- 73。
刘H,Kishimoto S,Yamamizo C,Fukuta N,Ohmiya A.类胡萝卜素积累和雌核生成基因表达的花瓣Eustoma羊藿.植物品种。2013;132(4):417 - 22所示。
- 74。
Livak KJ,Schmittgen TD。实时定量PCR的相对基因表达数据分析及2ΔΔCT方法。方法。2001; 25(4):402-8。
致谢
作者要感谢Md Sultan Mia博士将RNA-seq数据上传到NCBI SRA数据库的帮助。
资金
该研究由澳大利亚工业,创新和科学系全球创新联系计划(GIL53853)资助。资助机构没有参与数据的设计和收集,分析和数据的解释,或写作稿件。
作者信息
隶属关系
贡献
XW, HL和GY设计并构思了本研究。XW进行了实验。XW和HL进行了数据分析并撰写了手稿。KHMS和GY仔细审查了这篇文章,并提供了建设性的反馈。所有作者都通过了手稿的最终版本。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们在这项研究中没有竞争利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
具有值得注意的特性和位于XWMC153和XGWM155的目标QTL标记间隔内(物理位置为484,402,604-702,961,948 BP)。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
王旭东,刘海,西迪克,K.H.M.等等。小麦近等基因系的转录组分析显示,3A染色体上有抗收获前发芽的候选基因。BMC植物BIOL.21,53(2021)。https://doi.org/10.1186/s12870-021-02824-x
收到了:
接受:
发表:
关键词
- RNA测序
- 小麦
- 收获前发芽
- 标记辅助选择
- 近代线