摘要
背景
西伯利亚wildrye (Elymus sibiricus.在青藏高原草原建设和草场恢复中,由于其具有较强的抗逆性、较高的营养价值和易于栽培等优点而受到广泛的关注。然而,基因组信息的缺乏大肠sibiricus阻碍了其遗传学研究和育种过程。
结果
在这项研究中,我们进行了一个基因组调查并开发了一组SSR标记大肠sibiricus基于下一代测序(NGS)。我们生成了469.17 GB清洁序列,为6.86GB估计基因组大小的58.64倍。我们组装了4.34 GB的基因组草案,其重复元素为73.23%,杂合子比为0.01%和GC含量为45.68%。基于侏儒序列,我们确定了67,833个SSR基因座,从中随机选择四百个以开发标记。最后,30个标记在释放和十之间表现出多态性,被鉴定为单轨卡SSR。这些新开发的标记与先前报道的30种用于分析27野生的遗传多态性大肠sibiricus登记入册。我们发现单位点ssr的有效性优于多位点ssr。
结论
本研究提供了进一步全部基因组测序的见解大肠sibiricus在战略选择。新开发的SSR标记将为水稻的遗传研究和育种提供便利Elymus.物种。
背景
作为禾本科麦族中最大的一个属,Elymus.L.包含大约150种发生在世界上最温和的地区[1].由于它具有很好的抗旱、抗寒和抗病能力Elymus.物种可以为相关谷类作物的改良和育种提供重要的基因库[2].西伯利亚wildrye (Elymus sibiricus.L.),是一种模式种Elymus.属,多年生、凉季和自花传粉牧草[1].它广泛分布于欧亚大陆北部,因适应性强、耐寒、营养价值高、适口性好、易于栽培等特点,尤其在青藏高原草地建设和恢复中得到广泛应用[3.那4.]. 到目前为止,其种质资源特征、种内遗传多样性、系统进化和连锁图谱的构建越来越受到人们的重视[1那4.那5.].然而,与小麦、大麦等其他麦类谷物相比,该物种基因组序列的缺乏阻碍了其遗传学研究和育种的进展。
大肠sibiricus具有同种异体一体化基因组(ststhh,2n = 4x = 28)。平均核DNA含量(C值)大肠sibiricus通过流式细胞术(Fcm)确定为2c = 16.61pg,大约是来自属的可能的二倍体祖细胞的两倍Pseudoroegneria(stst)和大麦(HH) [6.].这种复杂性和巨大的基因组规模给全基因组测序带来了巨大的挑战大肠sibiricus.在一定的测序深度下,采用下一代测序(NGS)进行基因组调查是获得基因组大小、杂合度、GC和重复序列的另一种经济有效的方法。此外,基于基因组调查测序和硅质分析的分子标记的开发已成为遗传研究的实用工具[7.].
在各种类型的分子标记中,ssr (simple sequence repeats)具有多态性高、共显性遗传、重复性好、在基因组中分布广泛等优点[8.].这些特性在遗传图谱和指纹图谱构建、遗传多样性鉴定、分子标记辅助育种等遗传学研究中具有重要意义。利用基因组库或转录组序列和表达序列标签(ESTs)数据库的同源性搜索,可以开发出SSR标记。由于基因组转录部分的保守性,G-SSR (genomic DNA-derived SSRs)标记通常被认为比EST-SSR标记(EST-derived SSRs)具有更高的多态性。随着新一代测序技术(NGS)的不断发展,例如基因组调查测序,可以在经济上获取大量的基因组数据,并通过在大规模支架数据集中进行SSR基序搜索来进一步识别基因组SSR位点[9.那10那11那12].
一般来说,SSR被认为是位点特异性的,即单位点标记,因此,一对SSR引物只能扩增出一条或两条带(纯合子或杂合子)。然而,一对SSR引物除了获得期望的条带(单个位点)外,还经常获得复杂的条带(多个位点)[13].这可能是由于高等植物基因组大小大(特别是多倍体起源)以及基因组中重复DNA比例高,每个标记指向多个同源位点[9.那13那14].由于多位点SSR标记的存在,对包含特定基因的基因组位点的精确鉴定存在许多困难。例如,在一些多倍体物种的实际应用中,凝胶电泳的多位点SSR扩增产物可能来自多个基因组的多个位点,导致基因分型错误、多样性指数计算不准确等问题[15].相反,与多位点ssr相比,单位点ssr引物在基因组中定位独特,可为遗传研究和育种提供更可靠的基因型评分。
在本研究中,基因组测序适用于a大肠sibiricus品种“川草2号”采用Illumina Hiseq X-ten平台。第一个基因组草案大肠sibiricus在基因组调查数据的基础上,构建了一些单位点ssr和多位点ssr。此外,对27个野生种的遗传多样性和结构进行了分析大肠sibiricus利用这些新标记和之前发表的标记对材料进行鉴定。并对这些标记物的有效性进行了比较和评价。
结果
基因组测序和表征
14个270-bp的文库,对端为大肠sibiricus构建并测序。然后使用NCBI数据库对随机选取的10000对reads进行BLAST分析。BLAST结果表明小麦和大麦芽的近亲Elymus.,在所有库中均为最佳匹配种。此外,每个文库的读取与叶绿体基因组对齐大肠sibiricus结果显示匹配率低于5%,表明库建立无污染(数据未显示)。
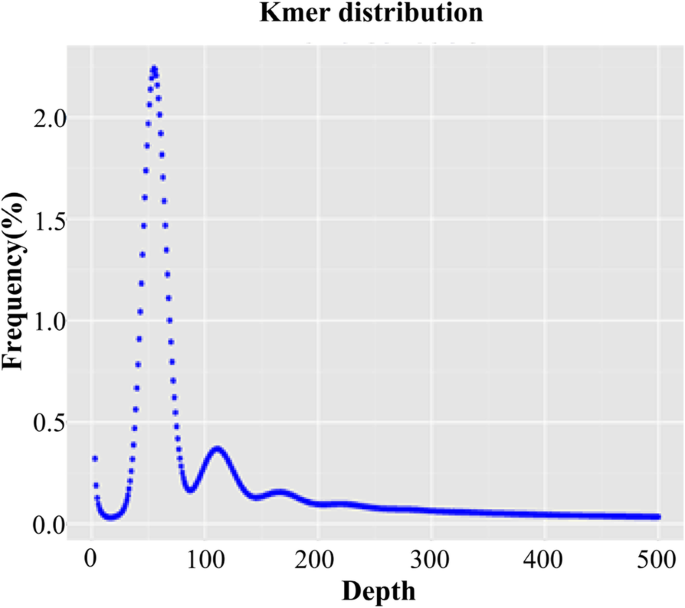
来自14个图书馆 使用Illumina-HiSeq X-ten平台生成了469.17gb的干净数据。估计的基因组大小大肠sibiricus大约6.86 gb,总测序深度为58.64折(表格1).对过滤后的序列进行高质量评分计算,Q20和Q30的百分比(测序错误率分别为1和0.1%)分别大于95.81和90.50%,表明测序过程的准确性较高(表11).然后对所有洁净数据进行25-mer (k = 25)频率分布分析。结果表明,k-mer深度分布峰值出现在54点(图1)。1).根据k-mer曲线分布计算,杂合度为0.01,重复序列占比为73.23%。
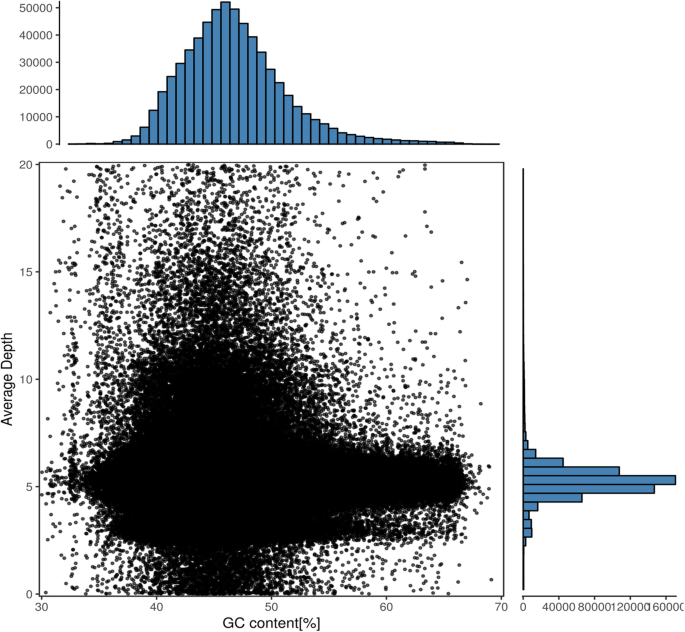
使用SOAP de novo程序进行de novo组装后,所有的clean reads总共产生了4,841,088 contigs, N50长度为2510bp,随后将其组装成支架(Table2).其中大于500 bp的序列为683040个,大于1 kb的序列为352851个,大于10 kb的序列为7536个。选择大于100bp的支架进行进一步分析。共生成了4,763,904个支架,N50为2648,大于500 bp、1 kb和10 kb的支架分别为684,597、352,659和8040 (Table .)2).组装的基因组草图约为4.34 Gb,占估计的6.86 Gb基因组的63.27%。计算GC含量为45.68%(表2)2),这与大于500 bp的支架构建的散点线图一致(图。2).共对25,993个基因进行了注释大肠sibiricus草拟基因组,每个基因平均转录本长度为2632.11 bp,平均编码序列长度为737.36 bp。预测每个基因的平均外显子数为4.72,每个转录本的平均外显子长度为311.32 bp(表S1).
使用RepeatMasker软件[16[我们注释了基因组草稿的重复区域,其中最丰富的重复元素是逆阻之温(16.45%),其次是DNA转座(3.26%)。逆压力和转座子中最常见的元素分别是长端子重复(LTR,15.90%)和TC1-IS630-POGO(0.25%)(表S.2).
全基因组SSR标记的开发
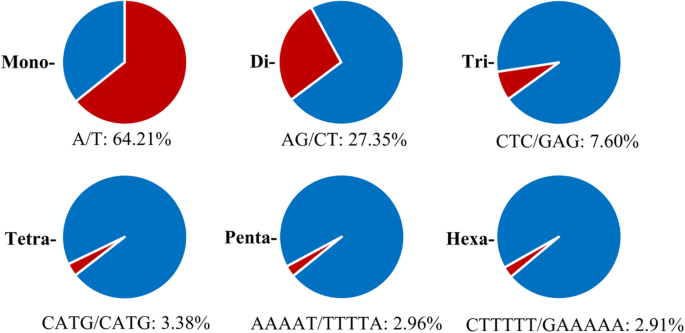
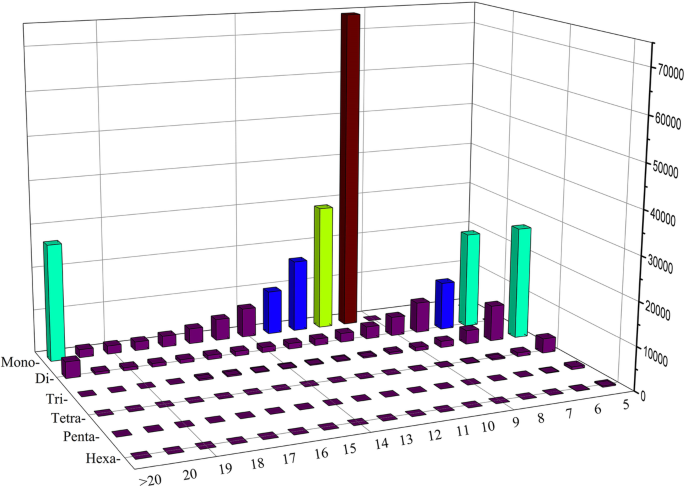
在507162(20.23%)个支架上搜索到约4.34gb的SSR位点,共获得315446个SSR。22611(0.90%)个序列含有一个在其他支架上未发现的位点SSR(表S)3.).在这些ssr位点中,单核苷酸、二核苷酸和三核苷酸基序的富集程度最高,分别占总ssr位点的61.81、21.59和14.55%(见表13.).核苷酸组成特征显示,A/T(64.21%)、AG/CT(27.35%)、CTC/GAG(7.6%)、CATG/CATG(3.38%)、AAAAT/TTTTA(2.96%)和CTTTTT/GAAAAA(2.91%)是单核苷酸-六核苷酸重复序列中最丰富的基元(图1)。3.).总体上,每种基元类型的基元重复数增加,基元丰度降低。4.).前三个丰富的图案重复数量为10,6和11,其SSR主题总数分别为77,930,31,980和31,575(表S4.).使用primer3软件[17],对上述22,611条含有SSR的单位点序列共设计了67833对SSR引物。在硅分析中,通过将这些引物的侧翼序列与基因组序列比对来验证这些引物的可靠性和多态性。随机选择400个SSR标记扩增8株植物的基因组DNA大肠sibiricus登记入册(表5.),其中30个为多态性,10个为单位点扩增子(ESGA-SL), 20个为多位点扩增子(ESGA-ML)(表S6.和无花果。1).
新SSR标记的多态性大肠sibiricus
标记多态性评价
总共60个SSR标记(30个ESGA,15 ES和15 ESG,表S6.)用于基因型27大肠sibiricus分别采自蒙古(MG)、青藏高原东部(QTP)和西伯利亚(SI)。10个ESGA-SL标记共产生29个等位基因(Na),平均为2.9个(表2)4.).所有ESGA-SL标记的平均多态性信息含量(PIC)值为0.391,其中ESGA-SL-7的PIC值最高,为0.595。观察杂合度的平均值(HO.)为0.480,其中ESGA-SL-10的H最高O.0.963。
在多位点标记中,ESGA-ML、ESGS和ES引物分别扩增出105、97和67条多态性条带,平均多态性百分比分别为90.52、82.49和73.84%(表S7.).与其他多位点标记(ESGS和ES)相比,新开发的ESGA-ML标记的PIC平均值(0.4059)和波段信息性平均值(BI, 0.6494)最高。曼惠特尼试验[18[表明,ESGA-SL和多基因座标记之间没有显着差异(表S.8.).对于MI、BI和Rp值,显著性差异(P. < 转录组发育的ES标记与基因组发育的ESGA-ML和ESGS标记之间的差异为0.01。
聚类与结构分析
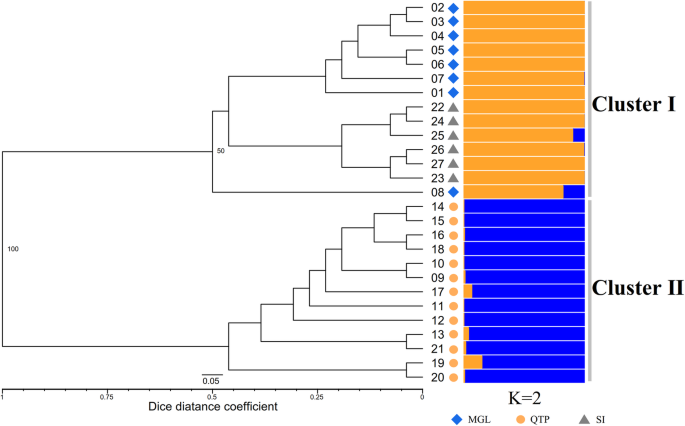
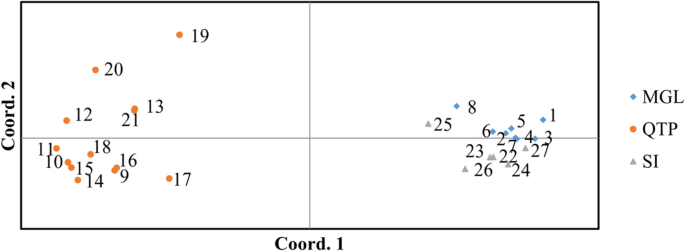
测试的27个人的基因成员大肠sibiricus基于ESGA-SL和ESGA-ML数据集的数据通过STRUCTURE 2.3.4软件获得(图1)。5.和无花果。2).结果显示出最佳的k值为2(k = 2),暗示测试所属的所属的遗传成员,其与使用ESGA-SL标记的UPGMA树木和主坐标分析(PCOA)一致。27种附加可以分为两个集群(群集I和群集II,图。5.和6.). 聚类Ⅰ包括所有蒙古(MG)和西伯利亚(SI)的种质,聚类Ⅱ包括所有东部QTP起源的种质。因此,通过UPGMA和PCoA分析,可以显著地鉴定出不同地区的野生种质,这表明基于ESGA-SL标记的野生种质具有很强的鉴别能力。然而,尽管来自不同地区的供试材料通过基于ESGA-ML标记的PCoA分析具有不同的特征(图3.),UMPGA树木图的拓扑结构含糊不清,无法区分27个野生载体(图。2).此外,基于ESGA-ML标记的UPGMA聚类和PCoA分析在ESGS标记中也发现了类似的结果(图S4.和无花果。5.).此外,与基因组开发的标记相比,转录组开发的ES标记不能很好地解释其结构隶属度(图S6.和无花果。7.).这表明G-SSR标记具有比用于内部分集分析的EST-SSR标记的卓越的辨别性。
的遗传多样性模式大肠sibiricus基于不同类型的标记
根据地理来源,将供试野生种质分为蒙古MGL、西伯利亚SI和青藏高原东部QTP三个地理类群。观察到的杂合度(HO.)的数据量从0.467变化到0.492(表1)5.).一般来说,N的值一种NE.Shannon信息指数(Shannon information index)、HE.ESGA-SL标记计算的各地理群的期望杂合度(expected heterozysity)和PP (Percentage of polymorphic loci)均高于ESGA-ML标记(Table5.).基于ESGA-SL和ESGA-ML标记和遗传分化系数(F圣)计算出来。结果表明,测试种质的遗传变异主要分布在具有中度遗传分化的地理群中(F.圣ESGA-SL和F圣ESGA-ML = 0.573)(表6.).
讨论
的特点大肠sibiricus草案基因组
下一代测序(NGS)的发展为研究人员提供了一种可实现的和更廉价的方法来获取植物基因组,特别是对非模式草物种,如Elymus sibiricus..对14个插入量为270 bp的文库进行Illumina测序,调查了该菌株的基因组草案大肠sibiricus在本研究中进行了从头测序和组装。草案基因组中GC含量适中(45.68%),说明Illumina平台很好地避免了测序偏倚。最终装配的contigs N50为2510 bp,远小于Lolium perenne.L.(contig n50 = 16,370 bp)[19],而略大于同质一百倍落花生hypogaeaL. (contig N50 = 696.6 bp) [20.].这可能是由于估算的基因组大小相对较大(6.86 Gb),重复性高(73.23%)大肠sibiricus以及库的短插入大小。估计的基因组大小大肠sibiricus(6.86 Gb)小于相关异源六倍体小麦(17 GB)[21],而比禾本科其他许多重要物种都要大,如大麦芽(5.1 GB)[22],山羊草属tauschii(4.5 Gb) (23],小麦属植物urartu(5.0 Gb) (24],Brachypodium distachyon(260 MB)[25],栽培稻(466 Mb)[26],高粱二色的(730 Mb) [27],Lolium perenne.(2 Gb) [28),Zea Mays.(2.3 Gb) (29].杂合性水平较低大肠sibiricus(0.01%)的k-mer分析可能是由自交交配系统引起的大肠sibiricus并指出其用于基因组测序的可行性。这是人类基因组的第一稿大肠sibiricus这对分子标记开发和功能基因挖掘具有重要意义。这项工作也为进一步利用更大的插入文库和新的测序技术如单分子实时测序提供了基础。
SSR标记的发展
SSR标记已广泛应用于遗传学研究和分子育种。在所有已识别的293,362 SSR中,广泛的SSR(97.95%)属于单核苷酸和三核苷酸基序,其类似于限制性位点相关的DNA-SEQ(RAD)的结果大肠高寒草场[30.].然而,在转录组测序研究中大肠sibiricus,三核苷酸基序具有最大数量[31],这可能是由于非编码和编码区中的序列之间的差异。通常,由于在选择压力下的三重密码子的富集,编码区具有更高的三核苷酸百分比[32].通常单子叶植物中最丰富的三核苷酸基序是CCG/CGG [33[虽然在这项研究中,这是CTC / GAG。这可能是不同物种中密码子使用偏差的结果[34].SSRS的A / T趋势大肠sibiricus也与报道的真核生物研究一致[35].每种motif类型的motif重复次数越多,motif丰度越低的现象与之前的研究一致[36].
对于多倍体物种,由于这些片段的相互重叠和不确定的等位基因,通常很难区分等位基因[37],这是很难进行基因型评分的。在这种情况下,单位点SSR标记被认为是最佳的选择,在大麦、花生和花生中均有报道丝瓜通过基因组调查[9.那12那38].本研究通过对黄花苜蓿基因组的调查,开发了10个单位点SSR标记大肠sibiricus在遗传变异研究和连锁图谱构建方面具有很大的应用潜力。
单位点和多位点标记的有效性比较
27野生的遗传多样性大肠sibiricus通过在本研究中开发的30个标记评估了加入,并在此前报告了其他30个。我们发现,在用聚丙烯酰胺凝胶分离时,通过在硅分析中筛选的预期单轨SSRS仍然表现出多基因座扩增子。这可能是由它们的侧翼序列的非保守性引起的[9.].通过对27个野生稻的遗传多样性分析,仅获得10个单位点(ESGA-SL)标记和20个多位点(ESGA-ML)标记大肠sibiricus登记入册。
10个ESGA-SL标记的平均扩增等位基因为2.9,其靠近同种异体四倍体物种落花生hypogaea(3.85)和芸苔属植物显著(3.23) (9.那37].10个ESGA-SL标记的PIC值在0.069 ~ 0.595之间,平均为0.391,表明其多态性丰富,具有较高的应用价值[39].ESGA-SL与其他3种标记系统(ESGA-ML、ES和ESGS)的PIC检测无显著差异,可能是单位点和多位点标记计算标准不同或单位点标记扩增位点有限所致。Mann Whitney检验表明,在PIC、MI和Rp方面,G-SSR (ESGA和ESGS标记)比EST-SSR (ES标记)更高效、多态,这可能是由于EST-SSR的侧序列更保守所致[40那41].此外,重要的(P.< 0.05) ESGA-ML标记的PIC值高于ESGS标记的PIC值,表明SSR标记开发方法测序优于传统方法。
基于ESGA-SL标记的UPGMA和PCoA聚类分析将27株野生稻进行了分类大肠sibiricus基于贝叶斯算法的结构分析也揭示了相同的模式。但其他3种多位点标记在揭示实际亲缘关系方面的能力均不如ESGA-SL标记。值得注意的是,所有的遗传多样性参数(N一种NE.,我,HE.和PP)均高于ESGA-ML,说明单位点标记揭示的遗传信息更准确,更适合于进一步的遗传分析[37].然而,略高的成对F圣根据ESGA-ML标记观察各组间的数值。由于多位点ssr在基因组定位上具有多个扩增位点的特点,不可避免地会覆盖一部分遗传信息。单基因座标记相对于多基因座标记的优势在本研究中得到了体现,然而,大量的单基因座标记覆盖了人类的整个基因组大肠sibiricus需要进一步确定或开发。在这种情况下,更高质量的全基因组测序和组装大肠sibiricus是必要的。
结论
在这项研究中,从头全基因组调查大肠sibiricus进行了4.34gB参考基因组序列,得到73.23%的重复元素,0.01%杂合子和45.68%GC含量。从基因组草案中鉴定了293,362个SSR标记,并通过Silico分析筛选了67,833个潜在标志物。随后,通过凝胶电泳验证了10个单轨道(ESGA-SL)标记和20个多基因座(ESGA-ML)标记,并在27中显示多态性大肠sibiricus登记入册。在遗传研究中,单位点标记比多位点标记具有更高的效率和信息量。基因组的研究和开发的SSR标记将为进一步的全基因组测序、分子育种和系统发育研究提供便利大肠sibiricus和相关的麦科物种。
方法
植物材料
大肠sibiricus四川草地科学院(Chengdu,China)提供的品种“Chuancao No.2”被流式细胞术鉴定为四倍体后被采用(图。8.)并种植在生长室(25℃,300μmols·m2·年代−116小时,光周期)。采用DNA提取试剂盒(中国北京天根)从新鲜嫩叶和干净叶片中分离“川草2号”的总基因组DNA。用生物光度计(Eppendorf,德国)检测DNA浓度和纯度,1%琼脂糖凝胶电泳检测质量。
文库建设和Illumina测序
根据说明书制备了14个插入量为270 bp的基因组对端文库,然后在Illumina HiSeq X-ten平台上进行测序。Clean reads按照以下过滤和校正准则获得[9.]:未识别核苷酸(N)少于10%;与适配器对齐的不超过10吨,最多允许10%的不匹配;最多有50%的碱基具有< 5的phred质量。排除了文库构建过程中PCR扩增过程中可能产生的PCR重复。此外,为了调查潜在的污染效应,我们随机选择了10000对clean reads,并使用BLAST在NCBI数据库中搜索[42].最后,对上述14个文库的核外DNA含量进行BLAST检测[43的叶绿体基因组大肠sibiricus(MK775250, 135075个基点)。
基因组组装、注释和鸟嘌呤+胞嘧啶(GC)含量分析
通过SOAPDENOVO2组装过滤的高质量序列[44]的k-mer size = 54,然后计算GC含量。采用同源性预测方法,对4个近缘种的基因组进行比对,对蛋白编码区进行识别,并进行基因预测。小麦[21],大麦芽[45],山羊草属tauschii[23),t . urartu[24, E-value cutoff设为1e-5。GeneWise软件[46]用于预测去除冗余后相应基因组区域的确切基因结构。最后,Trnascan SE软件[46]用于预测tRNA。
SSRs的识别和验证
对新获得的基因组序列集进行重复序列注释大肠sibiricus由RepeatMasker进行[16]追随Gramineae的重复序列数据库[47].然后perl5脚本微透露软件(http://pgrc.ipk-gatersleben.de/misa/)在基因组DNA序列中鉴定SSRs。识别标准为:单核苷酸重复次数≥8次;二、三、四、五、六核苷酸重复序列的数量均大于5 [48].引物设计参数设定:引物尺寸18 ~ 27bp,退火温度55 ~ 65℃,GC含量30 ~ 70%,最终产物长度100 ~ 300bp [17].在Silico分析中使用,设计的引物被映射回“Chuancao No.2”的装配序列,并且SSR与一个网站合并为潜在的单轨SSR [49].
基于遗传多样性的ssr评价大肠sibiricus种质资源
随机选择400对SSR标记进行合成,然后进行PCR和电泳筛选验证。只有0-2个扩增条带的引物被识别为单位点SSR,具有多态性的引物被命名为'大肠sibiricus基因组组件单个基因座(ESGA-SL)标记。类似地,同时具有多于2个扩增条带的引物被称为“大肠sibiricus基因组组装多位点标记(ESGA-ML)。此外,30对多位点标记包括15个G-SSR标记(ESGS [40),以及15对EST-SSR标记(ES [31)基于大肠sibiricus转录组也被选择去扩增相同的27个大肠sibiricus(表S5.,补充文件)。
扩增条带以基因型(单位点标记)或0/1二元矩阵(多位点标记)记录。用Freetree方法计算27份材料的Nei 's遗传距离(GD)矩阵[50,然后在Figtree中可视化UPGMA树状图[50]. 通过ntsysv2.2进行主坐标分析(PCoA)[51].结构v2.3.4 [52]进行了基于贝叶斯模型的遗传隶属度说明。采用混合料模型,将参数设置为5万老化和10万蒙特卡罗马尔科夫链(MCMC)。结构收割者[53然后施用]估计“最佳k”。分子方差(Amova)的分层分析使用Genalex进行[54]来计算等位基因的数量(N一种),有效等位基因(NE.),Shannon多样性指数(I),期望杂合度(HE.)、多态位点比例(PP)等遗传多样性参数。最后,观察到杂合度(HO.)和基因流动(nm)计算了单个位点ssr的数目。
数据和材料的可用性
支持本文结论的所有数据集都包含在本文及其附加文件中。原始DNA测序数据可在NCBI SRA数据库中获得,引文编号为“PRJNA680324”。
缩写
- ngs:
-
新一代测序
- QTP:
-
青藏高原
- FCM:
-
流式细胞术
- SSRs:
-
简单序列重复
- ests:
-
表达序列标签
- G-SSR:
-
基因组DNA衍生的SSRS
- ESGA-SL公司:
-
大肠sibiricus基因组组装单位点标记
- ESGA-ML:
-
大肠sibiricus基因组组件多基因座标记
工具书类
- 1.
关键词:麦胶蛋白,全球种质资源,遗传多样性,遗传多样性Elymus sibiricus..行罪学报。2009;18:59-66。
- 2.
杜威博士,巴克沃思我。与多年生妥塞氏植物分类的基因组分类系统。遗传。操纵植物品种。,proc。,int。Symp。1984; 35:202。
- 3.
严建军,白淑琴,马鑫,甘玉明,张建军。遗传多样性的Elymus sibiricus.以及它在中国的繁殖。[j] .植物学报,2007;24:226-31。
- 4.
引用本文马旭,张学强,周永红,白树青,刘伟Elymus sibiricus.(禾本科:麦科)青藏高原群体的ISSR标记研究。生物化学学报,2008;36:14 - 22。
- 5.
严建军,白淑琴,张新强,尤明明,张成斌,李德兴,曾燕。野生植物遗传多样性Elymus sibiricus.利用SRAP标记对中国青藏高原种质进行检测。草原学学报。2010;19:173-83。
- 6.
Vogel KP、Arumuganathan K、Jensen KB。小麦科多年生禾本科植物的核DNA含量。作物科学。1999;39:661–7.
- 7.
周伟,胡莹莹,隋泽华,傅飞,王建国,常丽萍,郭文辉,李炳柏。基因组调查、测序和遗传背景鉴定Gracilariopsis lemaneiformis(肾小序)基于下一代测序。Plos一个。2013; 8:E69909。
- 8.
江格。分子标记辅助育种:植物育种者的审查。Cham:Springer;2015. p。431-72。
- 9.
周XJ,东义,赵杰,黄L,仁XP,陈YN,黄SM,廖硕,雷Y,闫莉,江赫。基因组调查序列对花生单王子遗址SSR标记的开发和验证(落花生hypogaeal .)。BMC基因组学。2016;17:420。
- 10。
沙玛MK,沙玛R,曹pj,詹金斯J, Bartley LE, Qualls M, Grimwood J, Schmutz J, Rokhsar D, Ronald PC。柳枝稷基因组结构和组织的全基因组调查。《公共科学图书馆•综合》。2012;7:e33892。
- 11.
肖杰,赵杰,刘美娟,刘萍,戴玲,赵志辉。枣树SSR位点的全基因组鉴定及SSR引物的可移植性。《公共科学图书馆•综合》。2015;10:e127812。
- 12.
安建勇,尹明强,张强,龚德德,贾晓文,关玉军,胡军Luffa cylindrica微卫星高分辨率熔融(SSR-HRM)分析丝瓜基因型。int j mol sci。2017; 18:1942。
- 13.
Fluch S,Burg A,Kopecky D,Homolka A,Spiess N,Vendramin GG。挪威云杉可变SSR标记的特征(挪威云杉l .)。BMC Res. Notes. 2011;4:401。
- 14.
张强,李军,赵永波,科尔班,韩永平。利用SSR标记评价中国野生苹果品种和品种的遗传多样性Plant Mol Biol Rep. 2012; 30:539-46。
- 15.
陈淑琴,陈淑琴,陈淑琴,陈淑琴,陈淑琴,陈淑琴。相似起源的等位基因多样性的不同模式:以油菜为例(芸苔属植物显著在中国和澳大利亚。基因组。2008;51:1–10.
- 16.
Tempel S.使用和理解RepeatMasker。埃夫里市:胡玛纳出版社;2012.29-51页。
- 17.
Untergasser A, Cutcutache I, Koressaar T, Ye J, Faircloth BC, Remm M, Rozen SG。Primer3——新的功能和接口。核酸研究。2012;40:e115。
- 18.
Rahardja D,Zhao Yd,Qu Y. Wilcoxon-Mann-Whitney测试的样本大小测定:全面审查。生物制药研究中的统计数据。2009; 1:317-22。
- 19.
Byrne SL, Nagy I, Pfeifer M, Armstead I, Swain S, Studer B, Mayer K, Campbell JD, Czaban A, Hentrup S, Panitz F, Bendixen C, Hedegaard J, Caccamo M, Asp T.基于synteny的牧草基因组草案Lolium perenne.:用于细胞和分子生物学。植物J. 2015; 84:816-26。
- 20。
Bertioli DJ,Jenkins J,Clevenger J,Dudchenko O,Gao DC,Seijo G,Leal Bertioli SCM,Ren LH,Farmer AD,Pandey MK.异源四倍体花生的基因组序列落花生hypogaea.NAT Genet。2019; 51:877-84。
- 21。
关键词:面包小麦,六倍体,全基因组,全基因组小麦Gigascience公司。2017;6:1–7.
- 22。
Mascher M, Gundlach H, Himmelbach A, Beier S, Twardziok SO, Wicker T, Radchuk V, Dockter C, Hedley PE, Russell J.大麦基因组的染色体构象捕获有序序列。大自然。2017;544:427-33。
- 23。
赵国光,邹聪,李锴,王凯,李天宝,高丽芳,张学祥,王洪军,杨志军,刘学森山羊草属tauschii基因组揭示了转座子的多重影响。Nat植物。2017;3:946-55。
- 24.
凌总部、赵SC、刘DC、王JY、孙H、张C、樊HJ、李D、董LL、陶Y。小麦A基因组祖细胞的基因组草图小麦属植物urartu.大自然。2013;496:87 - 90。
- 25.
国际刷印倡议。模型草的基因组测序和分析Brachypodium distachyon. 自然。2010;463:763–8.
- 26.
国际水稻基因组测序项目。基于图的水稻基因组序列。大自然。2005;436:793 - 800。
- 27.
Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A. The高粱二色的基因组和草的多样性。大自然。2009;457:511-6。
- 28.
Byrne S, Panitz F, Hedegaard J, Bendixen C, Studer B, Farrell JD, Swain S, Armstead I, Caccamo M, Asp T.多年生黑麦草基因组测序(Lolium perenne.).国际植物与动物基因组大会XX。2011.
- 29.
Jiao YP、Peluso P、Shi JH、Liang T、Stitzer MC、Wang B、Campbell MS、Stein JC、Wei XH、Chin CS。利用单分子技术改良玉米参考基因组。自然。2017;546:524–7.
- 30.
刘荣军,卢晓伟,窦庆伟。SSR标记的开发大高寒草场基于简化表示的基因组测序。Mol Plant Breed. 2018; 16:1888-94。
- 31.
周Q,罗D,马LC,谢WG,王Y,王YR,刘ZP。西伯利亚野生黑麦EST-SSR标记的建立及其跨种转移性(Elymus sibiricus.L.)使用Illumina测序。科学。众议员2016;6:20549。
- 32。
Blanca J,Canizares J,Roig C,Ziarsolo P,Nuez F,Picób.转录组特征和高吞吐量SSR和SNPS发现Cucurbita浆果(葫芦科)。BMC基因组学。2011;12:104。
- 33。
Kantety RV, Rota ML, Matthews DE, Sorrells ME。大麦、玉米、水稻、高粱和小麦表达序列标签中简单序列重复的数据挖掘。植物学报,2002;
- 34。
秦泽,蔡泽,夏通克,王MC。同义密码子使用偏差与内含子数量相关,并且在植物中出现的外显子表现出不平衡。BMC基因组学。2013; 14:56。
- 35。
Tótg,Gáspáriz,jurkaj.微卫星在不同的真核基因组:调查和分析。Genome Res。2000; 10:967-81。
- 36.
林地草莓的基因组(草莓属vesca).NAT Genet。2011; 43:109-16。
- 37.
李海涛,尤纳斯M,王晓峰,李晓明,陈玲,赵波,陈旭,徐金生,侯峰,洪炳辉。异源四倍体油菜籽单位点SSR核心标记(芸苔属植物显著l .)。定理。达成。麝猫。2013;126:937-47。
- 38.
关键词:大麦,功能多样性,SSR, SNP,功能多样性摩尔品种。2008;22:1-13。
- 39.
利用限制性片段长度多态性构建人类遗传连锁图谱。Am J Hum Genet, 1980;32:314。
- 40.
雷逸涛,赵莹莹,于芳,李燕,窦庆伟。53个西伯利亚野生植物基因组- ssr多态性标记的开发与鉴定Elymus sibiricus.l .)。自然资源保护,2014;6:6 1 - 4。
- 41.
关键词:est,微卫星标记,小麦,a, B基因分型理论应用,2002;104:399-407。
- 42.
Altschul科幻。基本本地对齐搜索工具(BLAST)。中国生物医学工程杂志。1990;
- 43.
关键词:短寡核苷酸,序列比对,序列分析,序列分析生物信息学。2008;24:713-4。
- 44。
罗瑞波、刘波、谢依尔、李智、黄伟、袁杰、何光慈、陈义新、潘琦、刘义杰。SOAPdenovo2:一个经验上改进的内存效率的短读从头汇编程序。Gigascience公司。2012;1:18.
- 45。
国际大麦基因组测序联盟。大麦基因组的物理、遗传和功能序列组装。大自然。2012;491:711-6。
- 46。
tRNAscan-SE在线:整合搜索和上下文分析转移RNA基因。核酸杂志2016;44:W54-7。
- 47。
关键词:真核生物,重复元件,数据库,Repbase更新暴徒的DNA。2015; 6:11。
- 48.
利用EST数据库开发大麦基因衍生ssr标记(大麦芽l .)。定理。达成。麝猫。2003;106:411-22。
- 49.
舒勒GD。利用电子PCR进行序列定位。基因组研究》1997;7:541-50。
- 50.
关键词:毛滴虫,DNA指纹,系统发育树微生物学杂志。2001;51:731-5。
- 51.
Rohlf FJ。NTSYS-pc:数值分类和多元分析的微机程序。Stat。1987;41:330。
- 52.
Falush D, Stephens M, Pritchard JK。利用多位点基因型数据推断群体结构:显性标记和零等位基因。Mol Ecol Notes. 2007; 7:54 - 8。
- 53.
Earl DA, Vonholdt BM。结构收获器:一个可视化的结构输出和实现埃凡诺方法的网站和程序。2012; 4:359-61。
- 54.
Speall R,Smouse Pe。Genalex 6:Excel中的遗传分析。人口基因软件教学与研究。摩尔。ECOL。笔记。2006; 6:288-95。
确认
作者感谢NPGS(美国农业部国家植物种质系统)好心地提供了部分植物材料,即带有PI编号的材料。
资金
该工作得到了中国国家自然科学基金(3177131276)的支持,以及青藏高原高级饲料种质重点实验室(2020-ZJ-Y03)。这些融资机构没有进行数据的研究和收集,分析和解释和写作稿件。
作者信息
隶属关系
贡献
MX、WWD和LWH构思并设计了该作品;XY、XYL、DZX和YJ进行了实验;XY、LX和XYL对数据进行分析并撰写了本文。BSQ和YQQ对稿件进行了审核。所有作者都阅读并批准了原稿。
通讯作者
道德声明
伦理批准和同意参与
作者遵守了所有相关的机构,国家和国际指导方针。
同意出版物
不适用。
利益争夺
作者声明他们没有利益冲突。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充资料
附加文件1:表S1。
预测的基因信息大肠sibiricus以及它的相关物种。
附加文件2:表S2。
中重复元素的长度和比例大肠sibiricus.
附加文件3:表S3。
中SSRs的统计大肠sibiricus基因组。
附加文件4:表S4。
不同重复数SSR基序统计。
附加文件5:表S5。
野生大肠sibiricus本研究中使用的资料。
附加文件6:表S6。
本研究采用SSR引物。
附加文件7:表S7。
.多基因标记的遗传指标。
附加文件8:表S8。
不同标志物PIC、MI、BI、Rp的Mann-Whitney检验有统计学意义
附加文件9:图S1。
本研究单位点(ESGA-SL-2和ESGA-SL-9)和多位点(ESGA-ML-7和ESGA-ML-10)标记的PAGE电泳图示例。
附加文件10:图S2
. 研究了27个品种的UPGMA树状图和遗传结构大肠sibiricus基于ESGA-ML标记。
附加文件11:图S3
.对27例进行PcoA分析大肠sibiricus基于ESGA-ML标记。
附加文件12:图S4。
27学习的UPGMA树木和遗传结构大肠sibiricus基于ESGS标记。
附加文件13:图S5。
对27例进行PcoA分析大肠sibiricus基于ESGS标记。
附加文件14:图S6。
27学习的UPGMA树木和遗传结构大肠sibiricus基于ES标记。
附加文件15:图S7。
对27例进行PcoA分析大肠sibiricus基于ES标记。
附加文件16:图S8。
大肠sibiricus用流式细胞仪鉴定品种‘川草2号’为四倍体。红色是二倍体大麦芽L.,黄色是大肠sibiricus品种'Chuancao No.2'和粉红色是六倍体小麦.
权利和权限
开放获取本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非信用额度中另有规定。
关于这篇文章
引用这篇文章
熊毅,雷晓,白松。等。水稻基因组调查单位点和多位点SSR标记的测序、建立和鉴定Elymus sibiricus.lBMC植物杂志21,3(2021)。https://doi.org/10.1186/s12870-020-02770-0
收到了:
公认:
出版:
关键词
- 基因组测序调查
- Elymus sibiricus.l
- 标记的发展
- 单位点SSR
- 遗传多样性研究