摘要
背景
芸苔栗鸟是全球培养的重要油籽作物。在驯化和繁殖期间B. Napus.由于开花时间对产量有重大影响,因此一直是选择的目标。本研究采用双酶切限制性位点相关DNA测序(ddRAD)方法研究了水稻开花的遗传基础B. Napus..F2测绘群体来自早期开花的弹簧式和晚开花的冬季类型之间的交叉。
结果
在作图群体中,花期在个体间的差异可达25天。高基因型错误率在最初的质量控制后仍然存在,生物学测序重复之间的基因型不一致度为~ 12%。经基因型误差校正,构建了3981.31 cM、包含14630个单核苷酸多态性的连锁图谱。在C2染色体上检测到一个数量性状位点(QTL),包括8个花期基因方法.
结论
这些发现证明了ddRAD方法的有效性B. Napus.基因组。我们的结果还表明,DDRAD基因型错误率可以高于F的预期2人群。质量过滤和基因型校正和估算可以大大降低这些误差率并允许有效的联动映射和QTL分析。
背景
基因分型测序(GBS)是高通量发现作物基因多态性的强大工具[1,2,3.,4,5].GBS包括一系列图书馆准备和测序方法,其成本不同,有条不紊偏差和所产生的数据的类型和数量[1,6].限制性位点相关的DNA测序(Rad)是GBS方法,其可用于通过改变所用酶和测序深度来成本有效地校准基因分型基因座和单核苷酸多态性(SNP)的数量和覆盖。近期单一酶rad和两种酶双消化rad(ddrad)的比较分析在不同植物中使用了一系列酶组合,并表明Hinfi和HPyCh4iv的酶组合对于在一系列物种上最大化基因组覆盖范围[7].与其他GBS方法一样,DDRAD易于缺少数据和杂合基因型的欠息[5,但是输入和校正方法可以帮助产生高质量的基因型。作物中使用的归算和校正方法包括基于LB-Impute的隐马尔可夫模型[8]和fsfhap [9],基于滑动窗的基因型校正器[10.]简单的启发式方法[11.].
GBS已被用于标记发现,联动映射和QTL分析在一系列作物中[12.,13,14包括重要的油料作物芸苔栗鸟.已生成20多张高密度联动图B. Napus.使用RNA测序[15],芸苔属60k基因分型序列[16,17]和ddrad测序[18].结合表型数据,这些连锁图为农艺性状背后的基因鉴定提供了强有力的基础,这些基因可以被引入到作物种质中[19,20.].作物产量在B. Napus.对花期的依赖性很强,因此这一性状是一个关键的育种目标。在拟南芥中,开花时间的遗传途径已经被阐明,大多数开花时间基因在拟南芥和拟南芥之间是保守的B. Napus.[21,22,23].在此基础上,已检测到用于开花时间的许多QTL和相关的SNPB. Napus.[24,25,26,27,28,29,30.,31].然而,尽管在基因基础的理解上取得了进展B. Napus.开花时间,仍有大量的开花时间变异仍有待解释。
有三个B. Napus.油菜(OSR)生长类型,开花时间具有相当大的变异:春季,半冬季和冬季。Spring OSR和半冬季OSR对花卉的春化需求很低,并且正在开花,而冬季OSR具有强烈的春化要求,并且是延迟开花。在B. Napus.繁殖,春季OSR的开花性状与冬季OSR相比减少了生成时间,允许更多快速的育种周期。通过引入春季OSR等位基因来降低冬季OSR中的春化要求将是一种允许育种者加速冬季OSR育种的方法。此外,B. Napus.由于杂种优势,杂交品种通常比开放授粉的品种产量更高[32,33].如果可以有效地管理开花时间,可以从春季OSR和冬季OSR的杂交中利用杂种优势。因此,识别春季OSR和冬季OSR区分Spring OSR和冬季OSR的开花时间基因座具有重要的育种应用。在这里,为了识别这些基因座,我们跨过春季osr和冬季osr来生成f2映射人口。我们使用DDRAD测序进行基因组和亲本行。最后,我们构建了一种高密度联系地图,并进行了开花时间和相关性状萌芽时间的QTL分析。我们呈现开花时间和萌芽时间的候选地区,并讨论杂合育种群中的易于易于DDRAD基因分型的使用。
结果
预处理和比对测序读数
一个单独的个体(样本ID: 146)被排除在进一步的分析之外,因为它在修剪后只有不到100万个读取。在余下的206个样品中(包括199 F2(4个BnSOSR和3个BnWOSR),平均每个样本产生1314万条原始配对序列。平均有56.2%的读取是高质量的。覆盖碱基的平均覆盖深度为9.41 ×,基因组的平均覆盖广度为18.03%。
SNP滤波和基因型校正
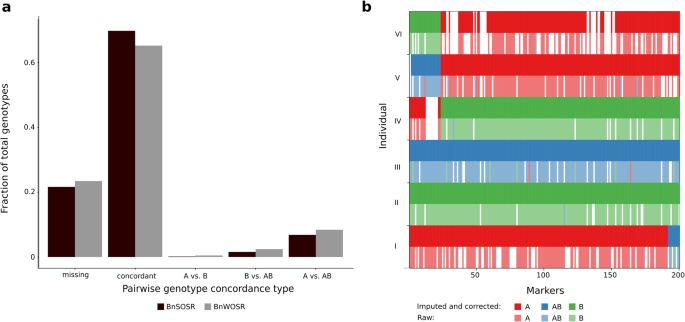
在绘图人群和父母个体中,共鉴定了4,841,931个双曲线SNP。为了进一步分析,七个父母单独被排除在SNP集中。通过个体缺失,基因型深度,次要等位基因频率(MAF)过滤,并且基因型缺失将SNP的数量降低至124,804。在过滤具有高基因型缺失的个体后保留了1999个后的192例。在124,804个SNP的中,50,856个在任何父母个体中没有杂合学基因型。患有父母个体中的杂合基因型的SNP可能是由父母基因组中的掩模或剩余的杂合子引起的,因此被排除在外。接下来,去除父母和5957之间的16,647个SNP,显示出偏析失真(p < 0.01), generated a set of 28,252 SNPs. Segregation distorted SNPs were distributed relatively evenly across chromosomes, with noticeable hotspots at the ends of chromosomes A1 and C5 (Fig.S1).基因型校正质量控制在过滤杂合子区域内的纯合子SNPs后进一步去除13509个SNPs。使用基因型校正器校正了4.94%的基因型,估算了94.76%的缺失基因型(图4)。1).最常见的基因型改正是B对AB(29.56%)和A对AB(23.48%)。
在亲本重复个体中,两两基因型一致性分析发现,平均基因型不一致性为12.28%(图2)。1).纯合基因型(A与B)之间的不间断罕见,纯合和杂合学基因型(A VS.AB,B Vs.Ab)之间的突出突出了97.51%的基因型不等调。
键映射
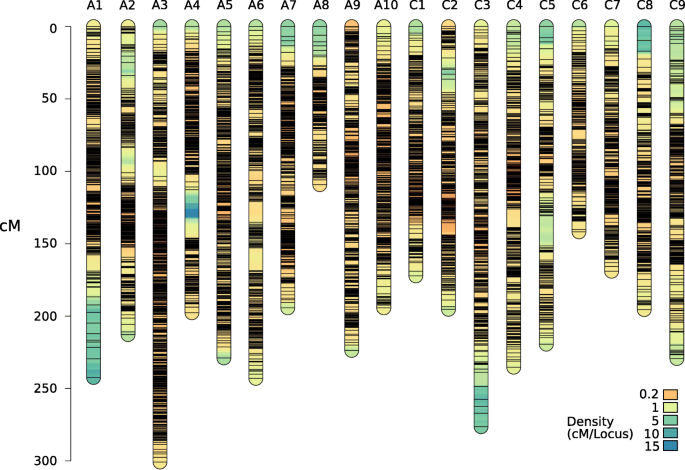
利用ASMap构建了3981.31 cM、包含14630个标记的连锁图谱(图1)。2和表格S1).基因组图谱为2147.15厘米,8587个标记物,C基因组图为1834.16厘米,具有6043个标记。在染色体A10上发现最高的平均标记密度,每MB的48.0标记。在基因组(28.33)上比C基因组(12.19)较高,每MB的平均标记密度较高。使用未经校正的标记构建补充图,其显示出遗传距离的高充气,总图长度超过30,000cm(图。S2).与用不同方法产生的六种公布的遗传图相比,纠正的遗传图仍显示出一些通货膨胀的指示(表S2).
遗传和物理地图顺序之间的相关性提供了关于遗传图和参考基因组之间的一致性的信息。平均Spearman的标记秩序每条染色体的秩相关为1.0(表1).观察到标记顺序的几个小不一致(图。S3).所有染色体的平均相关性均在0.98以上。平均每条染色体的个体交叉频率为2.79。
QTL映射
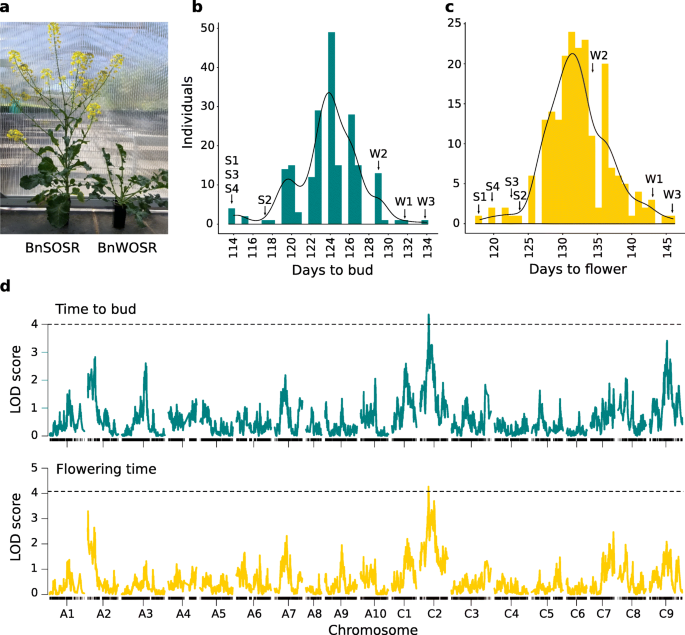
BnSOSR开花时间比冬季BnWOSR平均早20天(10 ~ 28天),出芽时间早17天(12 ~ 20天)S3).在F2子代,开花时间分布在亲本范围内(图。3.).单个显著(p< 0.05),在C2染色体上检测到性状开花时间和出芽时间的QTL区域重叠(图。3.).QTL的物理区域为开花时间20.57 Mb,出芽时间0.77 MbS4).开花时间QTL含有8个开花时间同源物,包括方法(表S5),萌芽时间QTL含有已知的开花时间同源物。携带BNWOSR等位基因在QTL峰值SNP导致芽和花的日子增加(图。S4).所鉴定的QTL对开花时间和出芽时间的表型方差解释率分别为9.08%和8.08%。在A2, A3和C9上也可以看到暗示性的LOD峰。
表型和QTL映射结果。一个播种后126天亲本个体发育对比照片。照片版权所有人为作者日月光。b绘制培养时间的群体表型的直方图。c花期群体表型定位直方图。BnSOSR的亲本个体S1-S4和BnWOSR的W1-W3。表中提供了表型数据S7.d花期和出芽期QTL定位的全基因组LOD图。重要的(p<0.05)基于置换测试的阈值,如虚线所示
低覆盖全基因组排序16早开花和19次延长开花F.2个体在C2上检测到137,696个变体。候选基因外显子的平均覆盖率为1.20倍(早开)和1.04×(晚开)(表S6).在候选基因编码序列中未观察到分离的非同义替换(表)S5),但在一个候选基因1 kb内发现3个分离的基因间变异(表S7).
讨论
优化基因分型逐序列策略
利用ddRAD进行基因分型,可获得14630个高质量的单核苷酸多态性,用于连锁和QTL定位。这项研究的发现可以帮助校准一系列研究目标所需的测序阅读和基因组位点的数量B. Napus.和相关的物种。对于GBS,研究人员通常旨在通过控制测序的reads与产生的位点数量的比例来优化基因组覆盖。在这里,基于HpyCH4IV和HinfI酶的硅消化,预期最大基因组覆盖宽度为24.6%。然而,观察到的平均基因组覆盖宽度较低,为18.03%。在拟南芥和拟南芥中使用HpyCH4IV和HinfI时,报告了更大的不一致甘氨酸最大,预期基因组覆盖率宽度为29.4和23.1%,但观察到的实验值分别为4.45和3.33%[7].这种不一致性被解释为片段大小选择偏差、硅基因座冗余和测序reads不足的产物[7].在B. Napus.在这里使用的人群中,决定覆盖率的最重要因素是每个样本的测序读取量。事实上,24个高测序量的样本显示,基因组覆盖广度超过预期的24.6%,达到最大值36.68%。这是特别令人惊讶的,因为覆盖率的计算只基于与高质量对齐的读取,这将大大减少覆盖率的宽度。这些发现表明,至少在B. Napus.,Igcoverage可以低估最大可实现的基因组覆盖范围。
使用HPYCH4IV和HINFI实现的高基因组覆盖范围表明这些酶非常适合于基因组多样性的高密度采样B. Napus..然而,当样本之间的测序工作不均衡时,高基因组覆盖广度会通过等位基因的缺失增加基因型的缺失。如果一个位点没有在足够深度的个体样本中进行足够的测序(这里的cut off是50%),它会在SNP调用或SNP过滤过程中被删除,成为一个缺失的基因型调用。高水平的缺失是简化表示排序的共同特征[3.]并且可以限制基因型数据在研究中不可能的研究的有用性[34].然而,如本研究所示,通过将避痛与基因组的高密度采样相结合,可以克服映射人群中基因型缺失的局限性。
键映射
物理图谱与遗传图谱的相关性较高,说明该图谱准确,与参考基因组共线。相似的,在早期的连锁图谱研究中也发现了只存在少量不一致的共线图谱B. Napus.[18,35].本文构建的连锁图谱在cM中平均比已发表的6个图谱大2倍B. Napus.使用具有相似性的标记密度的不同方法产生的联动地图[15,17,18,35,36,37].虽然我们的映射人群来自两个高度发散的父母,但是可以使我们能够采样比其他研究更多的交叉,但是一些残留的基因型误差或分离畸变预计会导致一些地图通胀。与我们的研究相比,依赖于f2群体,5个比较研究使用重组自交系或双单倍体群体,这可能会遭受较少的基因分型错误,因为它们内在缺乏杂合等位基因。使用基因分型阵列,比ddRAD基因分型更不容易出错B. Napus.F2人口也导致了一个更小的基因图谱,大约一半大小[17].这表明DDRAD基因分型错误,而不是人口类型,是遗传地图通胀的主要原因。然而,这里提出的联动地图的高密度和准确性表明它对于本地化QTL是有用的。
染色体上的开花时间QTL
开花时间QTLB. Napus.与拟南芥5号染色体同位的区域B. Napus.染色体A2, A3, A9, A10, C2和C3 [22].在C2上,我们鉴定了一个对开花时间和发芽时间有显著影响的基因座。这两个表型显著相关,可能与一个共享的QTL有关,可以解释~ 9%的变异。这种适度的变异是由分布在多个染色体上的许多位点控制的典型性状,每个位点对表型效应的贡献很小。此外,开花时间和出芽时间的QTL区域大小(LOD置信区间)也不同。QTL区域的大小很重要,因为它反映了所达到的作图分辨率水平。在F2绘制群体,QTL的置信区间可以大(> 1MB),并且这些群体通常代表候选基因的微映射的起点。在这项研究中,我们使用了1.5个单位的推荐LOD阈值为95%的置信区间覆盖[38].然而,置信区间的宽度取决于QTL峰值的陡峭,这可以取决于包括标记密度的一系列因子[39,40].
该QTL在物理上与之前的研究发现的一个位点很接近,该位点发现60 K阵列SNP bb - scaffold 18507_1-p889927与C2上v81位点33,936,984位(Darmor v41: 26,548,393位)的一个QTL相关,解释了6.36%的开花时间变异[[endnoty1]] [endnoty2]。28].但是,这里识别的QTL LOD峰值远离该位置。在C2上已知的开花时间基因中,FT.[41],方法[42),f[43]可能具有特别实质的效果。FT.已经牵连B. Napus.开花时间差异[30.,44),方法被发现可以解释约23%的开花时间变化B. Napus.[45].f是转录因子的抑制因子方法[43,46,但与本研究所鉴定的QTL区距离较远。的FT.C2的同源物位于QTL区域外1-3 Mb,在所有测试的生态型中均低水平表达[47],这表达了FT.也可能不是候选基因。候选的开花时间基因都没有显示出与开花时间隔离的非同义词或同义替换,表明监管变化可能是检测到的QTL。方法和SRR1.在QTL区域内,发现在其编码序列1 kb内存在变异,这些变异可能存在于早花和晚花F2个人。通过进一步研究候选基因的差异表达,有可能确定是否方法,SRR1.或者是另一个基因导致了开花时间的差异。总的来说,F2这里提出的实验设计只是发现过程的开始,因为紧密相连区域之间缺乏重组会阻碍高分辨率制图。我们的结果显示了一个显著的QTL和其他感兴趣的提示区域。这些位点中有几个可能需要转移到所需的遗传背景中,以利用春、冬品种之间的杂种优势。
基因型误差和校正
我们在重复的亲本样本中检测到高度的两两基因型不一致。由于在任何一个被比较的重复样本中,基因型错误都可能导致不一致,所以基因型错误率可以粗略估计为不一致的一半(~ 6%)。与GBS结果一致的是大米F2人口(11.,大多数错误可归因于杂合等位基因的低呼(97.51%)。亲本系是纯合子的,尽管残留杂合子性和错误定位可能导致杂合子等位基因调用。因此,后代的真实错误率可能比亲代高,因为F2群体含有50%的杂合子等位基因。
准确地呼叫杂合SNP,需要大大高于致电纯合SNP所需的深度[48,49].因此,本研究(9.41×)中使用的中等测序深度可以导致序列噪声的膨胀,并且等位基因采样不足,这可能导致杂合等位基因的损失[50].归因于杂合子的低估误差的百分比甚至可能是低估,因为明显纯合等位基因之间的误差可能是通过矛盾的杂合等位基因的错误基因型呼叫而引起的。
这里发现的基因型错误率高于文献中报道的错误率,即使是杂合子群体。例如,Malmberg等人(2018a)分析了一个杂合子B. Napus.使用不同的浏览序列和过滤的基因型呼叫使用最小读取深度的面板和过滤的基因型呼叫。作者发现2×测序覆盖范围的错误率为2.1%误差和1×测序覆盖的4.2%误差[50].同样,使用牛群中的GBS发现了3%的错误率,最小读取深度为5 [51].在一项利用慈鲷科鱼类作图群体进行的ddRAD基因分型研究中,基因型误差调查发现,在8 ×覆盖率时,错误率为4.41% [52].这表明GBS在F等样本中导致的基因型错误率高于预期2映射的人口。
发现了基因型误差对联系映射的影响。综合地图长度的累积膨胀通常由基因型误差引起寄生双重重组事件的基因型误差引起地图[53].此外,具有孤立失真的高水平缺失数据和标记也可能影响映射距离,因为这些改变了计算的重组率[54].据估计,标记中的每1%错误率大约增加了两个厘米到连杆地图[55].此前曾报道过小麦GBS数据的联动图通胀[53,54]和米[11.].在小麦的研究中,误差将联动地图膨胀至多11次[53].在大豆连锁及QTL定位研究中B. Rapa.基于GBS衍生的SNPs,错误率高(19.6%),得到的基因组连锁图谱膨胀,跨度4802.52 cM [56].
主要作物的连锁和QTL定位通常使用高度精确的商业基因分型阵列,如Illumina Infinium Brassica 60k阵列[57].基因分型阵列可能会引入抽样偏倚,因为它们只对已知的snp进行基因型。GBS相对于基因分型阵列的一个优势是,参考基因组中缺失的区域可以进行基因分型,并用于连锁图谱绘制。然而,基因分型阵列具有更准确地调用杂合子基因型和低缺失数据的重要优势。虽然基因型阵列也会产生经常涉及杂合位点的错误,但错误率可能较低,在1-2% [58,59].我们的研究结果表明,在杂合子群体中,基因分型阵列将产生比GBS的准确性显着更高的标记。这里,为了增加GBS数据的基因型精度,应用基因型校正。我们发现基因型校正显着降低了遗传映射膨胀,下调了使用GBS在低至中等测序覆盖范围内分析的杂合群体中的校正步骤的值。
结论
我们在C2上报告QTL,用于开花时间和萌芽时间B. Napus.冬天的类型x弹簧式十字架。该QTL和附加的提示位点可精细定位并回交于亲本品种中,以促进杂交春季型开花时间的控制x冬季型品种。在硅分析中还发现了一种优化的酶组合,由此产生的经验ddRAD位点和SNPs数量证明了酶组合HinfI和HpyCH4IV的有效性。此外,我们表明,ddRAD产生高水平的基因型错误,可以影响连锁图谱的构建。通过筛选SNPs,进行基因型分析和校正,构建精确的图谱,为花期的QTL分析提供依据。对控制开花时间和成熟的基因座的进一步研究将成为可能B. Napus.育种者更好地利用冬季和春季类型的变异。
方法
植物材料与表型
植物材料由BASF (Ghent)提供。这种栽培的植物材料不需要正式的鉴定。该作图群体由BASF的早春开花系(BnSOSR)和晚冬开花系(BnWOSR)杂交而成。F22017年,在珀斯的西澳大利亚大学(University of Western Australia, Perth)的一个植物管中,将由200个个体组成的种群以及4个BnSOSR和3个BnWOSR亲本复制直接播种到10x10x15厘米的花盆中。F2基于单植物调查人口。温度保持在18至22℃之间。为了确保发生开花,播种后63天开始春化,植物移动到受控环境室,恒定温度为4°C和8小时光周期。植物每周用两次浇水。大约6周后,播种后总共108天,植物返回植物。这6周的春化期是足够长的,以确保F的开花时间变化2常量反应的变异不驱动人口[60,61].每个壶都装有滴管,并与灌溉系统相连。植物每天浇水两次,每次1分钟。每2周人工提供含微量矿物质的肥料125 ml。记录了第一次花蕾出现的时间和第一次花开放的日期。所有的植物都要生长到种子结出为止。为了研究表型数据是否正态分布,使用ggpubr 0.2.1进行夏皮罗-威尔克检验并绘制直方图[62].
限制酶选择和消化
用两种限制性内切酶同时酶切DNA片段。根据反应缓冲液和培养温度的相容性,选择合适的酶对,产生粘性或悬垂末端,以允许同时消化。IgCoverage 1.0软件[7用于进行硅的消化B. Napus.darmorb -bzh v8.1 [63使用这些酶对。然后比较所选的26对限制性内切酶在100-600 bp大小范围内不同末端的片段数量(LengthDeFrag100-600),以及预期的基因组覆盖宽度百分比。在筛选出的26对适宜酶中,18对酶的覆盖广度> 20%(表1)S8).酶对HinfI和HpyCH4IV (New England Biolabs, Ipswich, USA)是根据每个样品的片段数量、基因组覆盖率、可用性和成本选择的。在100 ~ 600 bp范围内,预测产生840663个不同末端的片段,占基因组的24.6%。通过使用每个酶切酶5个单位和NEB CutSmart™缓冲液(10×) (New England Biolabs (NEB), Ipswich, USA)消化400 ng基因组DNA,确定所选限制性内切酶对的适用性。反应在37℃下孵育4小时,使用LabChip GX Touch 24 (PerkinElmer, Waltham, USA)显示结果。
适配器设计
DDRAD协议的适配器是根据Peterson等人使用的适配器和索引引物设计的。(2012)。修饰条形码适应剂以产生HPYCH4IV限制酶的互补突出,而常见的适配器被改变以为频繁切割寿命产生互补突出。如Peterson等人所述,通过退火10μm前向和反向链寡核苷酸组装。(2012)。使用Peterson等人描述的摩尔计算器确定用于条形码和公共适配器的连接步骤中的适配器浓度。(2012)。使用使用Igcoverage包获得的硅消化结果中的估计计算计算所需的限制性位点之间的平均距离。
测序图书馆准备
根据制造商的协议,使用DNeasy Plant Mini Kit (QIAGEN, Hilden, Germany)从叶子材料中提取基因组DNA。DNA浓度采用宽范围Qubit 3.0荧光测定法(Invitrogen公司,卡尔斯巴德公司,美国)进行定量,DNA质量采用LabChip GX Touch 24试剂盒(PerkinElmer公司,Waltham公司,美国)进行评估。使用Peterson等人(2012)和Clark等人(2014)协议的修改版本构建ddRAD库。提取的gDNA归一化浓度为50 ng/μL,酶切量为20 μL,酶切缓冲液为200 ng/ s。用HpyCH4IV (5u)和Hinfl (5u)在NEB CutSmart™缓冲液中进行消化,制备ddRAD库。37℃孵育反应4 h。
使用T4 DNA连接酶(Thermo Invitrogen,Carlsbad,USA)分别将消化的DNA连接到独特的条形码和普通衔接子。18μl含有连接缓冲液的18μl主混合物,200u的T4连接酶和普通适配器,并直接加入到20μL消化反应中,之后加入单独的条形码适配器。将反应物在22℃下孵育2小时,然后将65℃孵育20分钟,然后以每90℃的2℃的斜率冷却至4℃。为了适应DNA浓度和质量的变化,在连接后未合并样品,但是选择单独纯化和选择的双尺寸以实现250-800bp之间的碎片。通过加入60μl核酸酶的游离水将样品的总体积调节至100μl。通过将50μl1:4(0.5x)混合物添加到PEG缓冲液(20%PEG W / V,2.5M NaCl)中加入50μl的Ampure XP珠粒(Beckman Coulter,Brea,USA)来进行双尺寸选择以除去碎片> 800 bp。将上清液转移至20μL的1:1(0.7×)Ampure XP珠珠,以收集碎片> 250bp。使用80%乙醇洗涤珠子,并在30μl核酸酶的碎片中洗脱。
取10 μL大小的DNA进行PCR扩增。制备了40 μL的Phusion Hot-Start high fidelity master mix Polymerase (Thermo Fisher Scientific, Walthan, USA)主混合、正向(0.5 μM)引物和反向引物。样品在98°C扩增2 min,然后98°C扩增15 s, 62°C扩增30 s, 72°C扩增30 s,最后在72°C扩增5 min。使用1.5X Ampure XP Beads对扩增文库进行清洗,以去除引物二聚体。使用高灵敏度(HS) Qubit 3.0荧光测定法测定文库DNA浓度。使用LabChip GX Touch 24可视化库质量和片段大小分布。将等摩尔量的文库汇集在1.5%琼脂糖凝胶上,富集并选择300 ~ 700 bp的片段。使用QIAquick凝胶提取试剂盒(QIAGEN, Hilden, Germany)提取DNA。最终文库浓度、质量和大小分布再次评估,并使用10 nM Tris Buffer (pH 8.5, 0.1%吐温20,10 nM)调整至20 nM DNA。最终的文库被送往加文医学研究所的KCCG核心实验室,在HiSeq X Ten平台上进行配对末端测序。
为了帮助检测潜在的因果变量,有助于在f中开花时间2在低盖度下,16个开花时间小于127天的个体和19个开花时间超过137天的个体进行了测序S9).如上所述提取用于低覆盖的基因组DNA全基因组测序。根据制造商提供的指南,使用IlluminaTruseq®NanoDNA文库预备套件制备测序文库。汇集了Equimolar数量的唯一索引库,并在澳大利亚基因组研究设施的Hiseq 2500平台上发送成对结束排序。
适配器修边和质量修边
Illumina bcl2fastq 2.20.0.422管道[64]用于将基本呼叫文件转换为FASTQ格式。配对端DDRAD测序使用Saber 1.0解复用[65],只允许单次不匹配。原始FASTQ文件用Trimmomatic 0.36裁剪适配器序列和低质量的基础[66].对于适配器修剪,将适配器序列的最大不匹配分数用于适配器序列,以及30的回文夹得分阈值和10的简单剪辑得分阈值。从一开始,修剪低质量+ 33得分的低质量碱基并结束阅读。使用4底宽窗口进行滑动窗帘,当每碱平的平均质量低于15时,将基座修剪。提供带Trimmomatic的Illumina Truseq-PE适配器列表用于适配器修剪。丢弃修剪后少于36个碱基的所有读数都被丢弃。修剪后未配对的读数也被丢弃。读取预处理后,使用诊断工具FASTQC分析未经监控和修剪的读取[67].然后使用multiQC对每个样品的快速qc结果进行汇总和总结[68].MultiQc报告用于验证是否已删除适配器并读取质量很高。
对齐测序读取
使用BWA-MEM算法使用BWA 0.7.17映射修剪读取[69]B. Napus.darmo -bzh v8.1程序集[63]使用默认参数。对齐后,SAM文件将使用samtools 1.8转换为BAM格式[70].丢弃未映射的读取,补充对齐和具有映射质量的读取的读数被丢弃。此过滤器删除多映射读取,通常发生B. Napus.由于其多倍体基因组的同源区域。使用SAMtools stats和mosdepth 0.2.3分析制图结果[71].使用BedTools 2.26.0从MOSDepth PER-BASE输出计算DDRAD基因座的数量[72]在100 BP之内合并邻近的基因座。
称为单核苷酸变异
使用GATK 3.6调用变体[73].首先,使用SAMtools对BAM比对进行索引,然后使用HaplotypeCaller为每个样本调用snp。基因分型使用GATK GenotypeGVCF进行,使用默认设置,在读取棒禁用时自动创建索引并锁定。结果采用GATK catvariant合并每条染色体。使用VCFtools 0.1.15过滤变异[74].排除了Indels和多等位snp(-- remove-indels——max-alleles 2——min-alleles 2).在过滤SNP之前,除去具有> 0.9缺失基因型的个体。为了减少由于读取深度不足而被称为纯合等位基因的杂合等位基因的速率,具有深度<5的基因型<5(−-minDP 5)被认定为失踪。如果snp显示的次要等位基因频率< 0.05 (−加0.05)或当基因型不存在于所有个人的80%(−-max-missing 0.8).采用snpEff 4.3 t计算基因型不一致性[75]使用双亲的重复样本(弹簧型BnSOSRn= 4,冬天的类型BnWOSR withn = 3) with pairwise comparisons of genotypes for SNPs passing the above filters. Heterozygosity per individual was calculated using VCFtools.
使用自定义脚本vcf2gt.py进行扭曲snp的亲子分配和过滤[76],使用cyvcf2 0.8.0 [77]解析VCF文件。在Scipy 1.2.0中实施的Chi-Square测试[78进行了识别和丢弃具有显着隔离失真的SNP(p < 0.01) based on the expected F2分离比例为1:2:1。进一步过滤除去在至少一个父母中杂合的SNP,或者在父母之间不存在多态。该脚本还将VCF格式的SNP转换为AB格式的基因型矩阵(A:来自父母1的纯合等位基因; B:来自父母2的纯合等位基因; AB:杂合等位基因; - :缺少等位基因)。
使用gene - type- corrector 1.0输入和校正基因型[10.],非常适合F2中度到低基因型缺失的人群,就像这里分析的。该软件利用基因组参考上的SNPs顺序和滑动窗口方法,根据F2人群。在校正前,将150 bp基因组区间内最多8个连续的纯合子SNPs与基因型- corrector qc_hetero合并成一个单一SNP。这有助于在使用滑动窗口方法时防止杂合基因型对纯合基因型的错误校正。由于这里使用了20%的缺失SNPs,根据作物定位群体的经验测试,基因型校正器的预期准确率为> 95% [10.].
键映射
使用MSTMap算法进行联动映射[79]在R包ASMap中实现[80].利用重组事件和目标函数寻找最优基因座序列,并进行遗传算法优化p-Value阈值将群集标记分为连锁组被设置为1E- 23.基于评估1e的一系列值- 14.到1E.- 29..利用kosambi距离函数估计SNPs之间的遗传距离,将罕见的重组事件作为错误处理(detectBadData = True).根据参考基因组上的标记位置对连锁组进行染色体命名。不代表整个染色体的小连锁群与染色体连锁群合并,如果使用物理标记位置能够明确分配,则重新计算遗传距离。标记< 7的连锁组被丢弃。利用rqtl函数plotRF计算标记间估计的两两重组片段[38].利用重组片段识别不属于LD的离群标记,并利用物理标记位置手动修正标记顺序。在进一步分析中,共去除95个异常标记。为了确定遗传图谱的质量,使用r中的Spearman秩相关检验计算遗传图谱上标记顺序与参考基因组之间的相关性。交叉频率使用rqtl函数locateXO和自定义python脚本cross .py [76].
QTL映射
利用rqtl scanone进行QTL定位,利用单个QTL模型和开花和出芽时间非参数QTL模型进行QTL定位,因为这些性状不服从正态分布。使用1000次迭代的排列检验估计了对比值评分(LOD)的全基因组显著性阈值[81].使用RQTL LODINT估计每个QTL位置的1.5卢氏液滴间隔。使用具有Haley-knott回归的RQTL FITQTL来计算为每个QTL解释的方差百分比。用于rstudio 1.1.456中的QTL映射的自定义交互式R脚本[82]是基于RQTL手册[38].
花期基因的鉴定
来自开花交互数据库的总共306个拟南芥开花时间(FT)基因[83]下载自拟南芥资讯资源[84].这些基因包括已知的同源物B. Napus.开花时间的基因。爆炸+ 2.2.29 [85,86进行了对参考基因组的FT基因的分析,以使用1E的切断来找到基因同源物−6(下列的 [87])。使用BEDtools合并重叠的点击。v81注释中的基因名称[63],利用BEDtools获得与BLAST比对重叠的基因注释。
早花期和晚花期的变异分析2全基因组测序覆盖率低的个体
读取被对齐和使用上面描述的方法称为DDRAD测序数据的变体。为了帮助检测潜在的因果变量,应用了不太严格的VCFTOOLS滤波器,以排除具有高缺失或低次次次所有内部等位基因的变体(−-max-missing 0.25——mac 5).为了检测晚花期和早花期样本之间分离的候选变异,我们使用了一组简单的阈值。首先,我们要求两组每组有四个或更多的基因型调用。其次,我们要求每组中70%以上的基因型调用是一致的。最后,共识的基因型呼叫必须在不同组之间有所不同。
可用性数据和材料
所有序列均沉积于SRA (https://www.ncbi.nlm.nih.gov/bioproject/prjna640838.)在表中列出了个别换乘S10.用于本研究的脚本可在以下网站获得https://github.com/ascheben/bn_gbs/.基因型数据可通过figshare (https://doi.org/10.6084/m9.figshare.13139378.v1).本研究中使用的植物材料可从巴斯夫获得,但由于其可获得性受到限制,因此无法公开获得。
缩写
- RAD:
-
限制性位点相关的DNA测序
- ddRAD:
-
双消化RAD
- GBS:
-
Genotyping-by-sequencing
- LOD:
-
概率的对数
- VCF:
-
变体电话格式
- QTL:
-
数量性状位点
- SNP:
-
单核苷酸多态性
- 加:
-
轻微的等位基因频率
- osr:
-
油菜油菜
- WOSR:
-
冬季osr.
- SOSR:
-
夏季奥斯尔
参考文献
- 1。
K. R. Andrews, J. M. Good, M. R. Miller, G. Luikart, P. A. Hohenlohe(2016)利用RADseq的力量进行生态和进化基因组学研究。Nat Rev Genet 17,81 - 92。https://doi.org/10.1038/nrg.2015.28.
- 2.
K. Voss-Fels, R. J. Snowdon(2016)通过高分辨率基因分型了解和利用作物基因组多样性。植物营养与生物工程学报14(3),513 - 513。https://doi.org/10.1111/pbi.12456.
- 3.
J. A. Poland, T. W. Rife(2012)植物育种和遗传学的基因分型测序。植物基因组5,92-102。https://doi.org/10.3835/plantgenome2012.05.0005.
- 4.
S. Deschamps, V. Llaca, g.d. May(2012)植物基因分型。生物1,460 - 483。https://doi.org/10.3390/biology1030460.
- 5.
A. Scheben,J.Batley,D. Edwards(2017)基因分型逐个测序方法来表征作物基因组:选择合适的应用程序的正确工具。工厂Biotech J 15,149-161。https://doi.org/10.1111/pbi.12645.
- 6.
Xu X., Bai G.(2015)全基因组重测序:改变SNP检测、分子定位和基因发现的范式。摩尔品种35,33-33。https://doi.org/10.1007/S11032-015-0240-6.
- 7.
Y. B.Fu,G.W.Peterson,Y. Dong(2016)增加基因组采样和改善基因分型的SNP基因分型,并以新的限制酶组合进行逐序列。基因基因类型。6,845。https://doi.org/10.1534/g3.115.025775.
- 8.
C. A.Fragoso,C. Heffelfinger,H. Y.Zhao,S.L.Dellaporta(2016)从低覆盖序列数据中冒险在双层群中的基因型。Genetics 202,487-495。https://doi.org/10.1534/genetics.115.182071.
- 9.
K. Swarts, H. Li, J. A. Romero Navarro, D. An, M. C. Romay, S. Hearne, C. Acharya, J. C. Glaubitz, S. Mitchell, R. J. Elshire (2014) Novel methods to optimize genotypic imputation for low-coverage, next-generation sequence data in crop plants. Plant Genome 7, 1–12.https://doi.org/10.3835/plantgenome2014.05.0023.
- 10。
C. Miao,J. Fang,D. Li,P. Liang,X. Zhang,J. yang,J.C.C.C.C.C.C.Cabnable,H. Tang(2018)基因型校正器:改进的基因型调用F2和RIL种群的遗传绘图。SCI REP 8,10088。https://doi.org/10.1038/s41598-018-28294-0.
- 11.
T. Furuta,M. Ashikari,K.K.Jena,K. Doi,S.Reuscher(2017)适应米F2种群的基因分型逐序列。基因基因类型。7,881-893。https://doi.org/10.1534/g3.116.038190.
- 12.
J.G.uitdekigen,A.M.Wolters,B.D'Hoop,T.J.Borm,R.G.Visser,H.G.Van Eeck(2013)对高杂合子自身传递物型逐序列的基因分型逐序列测序的下一代测序方法。Plos一个8,E62355。https://doi.org/10.1371/journal.pone.0062355..
- 13.
H. Sonah, L. O’donoughue, E. Cober, I. Rajcan, F. Belzile(2015)利用GBS-GWAS方法鉴定大豆8个农艺性状的位点并进行QTL定位验证。植物营养与生物工程学报13(3),201 - 201。https://doi.org/10.1111/pbi.12249.
- 14.
m . m .只是l . w . Pembleton r . c .柏丽m·c·德雷顿s Sudheesh考尔,h . Shinozuka p Verma Spangenberg g . c, h·d·Daetwyler et al。(2018)通过转录组Genotyping-by-sequencing:实现在各种作物物种不同生殖习惯和倍性水平。植物营养与肥料学报,16(6):741 - 744。https://doi.org/10.1111/pbi.12835.
- 15.
C. Bancroft, C. Morgan, F. Fraser, J. Higgins, R. Wells, L. Clissold, D. Baker, Y. Long, J. L.孟,x.w Wang等.(2011)通过转录组测序分析多倍体作物油菜的基因组。Nat Biotechnol 29, 762-766。https://doi.org/10.1038/nbt.1926.
- 16.
李斌,高建军,陈建军,王振华,沈伟,易斌,文俊,马超,沈建军,傅涛等.(2019)控股分支的主要基因座的识别和精细映射芸苔栗鸟.定理:麝猫。https://doi.org/10.1007/s00122-019-03506-x.
- 17.
F. Sun,J. Liu,W. Hua,X. Sun,X. Wang,H. Wang(2016)通过组合联系和关联映射识别种子油含量的稳定QTLS芸苔栗鸟.植物科学通报252,388-399。https://doi.org/10.1016/j.plantsci.2016.09.001.
- 18.
陈晓霞,李晓霞,张斌,徐建军,吴振华,王斌,李海华,尤纳斯,黄磊,罗永强等.(2013)用伪参考序列的多倍体作物中限制性片段相关多态性的检测和基因分型:同种异体四倍体的案例研究芸苔栗鸟.BMC基因组学14,346-346。https://doi.org/10.1186/1471-2164-14-346.
- 19.
A. Abe, S. Kosugi, K. Yoshida, S. Natsume, H. Takagi, H. Kanzaki, H. Matsumura, K. Yoshida, C. Mitsuoka, M. Tamiru等.(2012)基因组测序揭示了使用Mutmap中的大米中的复杂重要基因座。NAT Biotechnol 30,174-178。https://doi.org/10.1038/nbt.2095.
- 20。
D. Edwards, J. Batley, R. J. Snowdon(2013)利用下一代测序技术获取复杂的作物基因组。Theor Appl Genet 126 1-11。https://doi.org/10.1007/S00122-012-1964-X..
- 21。
M.Tadege,C.谢尔顿,C. A. Helliwell,P. Stoutjesdijk,E. S. Dennis,W. J.孔雀(2001)控制开花时间方法orthologues在芸苔栗鸟.工厂J 28, 545-553。https://doi.org/10.1046/j.1365-313x.2001.01182.x..
- 22。
I. A. Parkin,S. M. Gulden,A.G.Sharpe,L.Lukens,M. Trucks,T.C. Osborn,D. J. Lydiate(2005)分段结构芸苔栗鸟基于比较分析的基因组拟南芥.遗传171年,765 - 781。https://doi.org/10.1534/genetics.105.042093.
- 23。
L. S. Robert,F. Robson,A. Sharpe,D. Lydiate,G. Z:1998)保守的结构和拟南芥开花时间基因的功能君士坦斯在芸苔栗鸟.植物分子生物学37,763-772。https://doi.org/10.1023/A:1006064514311.
- 24.
H. J.坚A.十张,J. Q.马,T. Y.王,杨B.,L。S.爽,刘M.,J. N.李,十楼旭,A. H.帕特森等人.(2019)联合QTL定位和转录组测序分析揭示了拟南芥开花时间基因芸苔栗鸟L. BMC基因组学20,21。https://doi.org/10.1186/s12864-018-5356-8.
- 25.
李宝军,赵文刚,李大仁,超海波,赵小平,塔南,李永华,关志斌,郭立贤,张丽娜等.(2018)基于环境稳定性和特异性QTL的开花时间机制的遗传解析芸苔栗鸟.植物科学学报277,296-310。https://doi.org/10.1016/j.plantsci.2018.10.005.
- 26.
等。关键词:大数据,大数据,大数据引用本文.(2013)油菜开花时间位点的遗传和物理定位(芸苔栗鸟l .)。Theor Appl Genet 126, 119-132。https://doi.org/10.1007/s00122-012-1966-8.
- 27.
(in chinese)沈永胜,向阳,徐恩生,葛晓辉,李志勇(2018)外源基因导入衍生的株高、分枝起始高度、茎粗和开花时间的主要共定位QTL芸苔栗鸟DH群体。植物科学学报9,392。https://doi.org/10.3389/fpls.2018.00390.
- 28.
L. P. Xu,K。胡锦涛,Z. Q. Z. Zhang,C. Y. Guan,S. Chen,W. Hua,J. N.Li,J.Wen,B. yi,J. yi,J. X. yi,J. X. yi,J. X. yi,J. X. yi,J. X. yi,J. X. Yi,J. X. Yi,J. X. yi,J. X. Yi,J. X. yi,J. X. Shen等.(2016)基因组 - 范围协会研究揭示了油菜籽的开花时间的遗传建筑(芸苔栗鸟l .)。DNA Res 23, 43-52。https://doi.org/10.1093/dnares/dsv035.
- 29.
M. N.Nelson,R.Rajasekaran,A. Smith,S. Chen,C.P.Peeck,K.H.M. Siddique,W. A. Cowling(2014)夏季每年发现的热时间进行热时间的定量特质基因座芸苔栗鸟L. PLoS One 9, e102611。https://doi.org/10.1371/journal.pone.0102611.
- 30.
H. Raman, R. Raman, Y. Qiu, A. S. Yadav, S. Sureshkumar, L. Borg, M. Rohan, D. Wheeler, O. Owen, I. Menz等.(2019) GWAS提示开花位点T在油菜花期和产量相关性状中的多效性作用。BMC基因组学20,636。https://doi.org/10.1186/s12864-019-5964-y.
- 31.
Osborn TC, Kole C, Parkin IA, Sharpe AG, Kuiper M, Lydiate DJ, Trick M芸苔属植物拉伯,B显著和拟南芥.遗传学。1997年; 146:1123-9。
- 32。
K. P. Starmer, J. Brown, J. B. Davis(1998)在爱达荷州北部种植的春季油菜杂种优势。作物科学38,376-380。https://doi.org/10.2135/cropsci1998.0011183x003800020018x..
- 33。
H. Cutforth, B. McConkey, S. Brandt, Y. Gan, G. Lafond, S. Angadi, D. Judiesch(2009)加拿大半干旱草原肥料氮响应与油菜产量。中国植物科学(英文版)89,513 - 513。https://doi.org/10.4141/Cjps08128.
- 34。
H. alipour,G.H.Bai,G. R. Zhang,M.R. Bihamta,V.Mohammadi,S. A.Peyghambari(2019年)使用大麦和小麦基因组参考的小麦基因分型逐序列(GBS)数据的注重准确性。Plos一个14,E0208614。https://doi.org/10.1371/journal.pone.0208614.
- 35。
(in chinese)中国水稻无瓣性状的QTL定位与QTL构建芸苔栗鸟植物科学6,1164。https://doi.org/10.3389/fpls.2015.01164.
- 36.
W. Ecke, A. Kampouridis, K. Ziese-Kubon, A. C. Hirsch(2015)利用高通量SNP分析油菜品种间替代系的鉴定和遗传特性(芸苔栗鸟(L.)具有增强的胚胎发生潜力。Theor Appl Genet 128, 587-603。https://doi.org/10.1007/S00122-015-2455-7..
- 37.
W. E. Clarke, E. E. Higgins, J. Plieske, R. Wieseke, C. Sidebottom, Y. Khedikar, J. Batley, D. Edwards, J. L.孟,R. Y. Li等.(2016)高密度SNP基因分型阵列芸苔栗鸟基于异源四倍体基因组单位点标记的优化选择及其祖先二倍体物种。Theor应用Genet 129, 1887-1899。https://doi.org/10.1007/s00122-016-2746-7.
- 38.
(in 2003) R/QTL:试验杂交组合的QTL定位。生物信息学19日889 - 890。https://doi.org/10.1093/bioinformatics/btg112.
- 39.
利用一组密集标记定位数量性状位点的统计方法。遗传学。1999;151:373 - 86。
- 40.
Mangin B,Goffinet B,Rebai A.构建QTL位置的置信区间。遗传学。1994年; 138:1301-8。
- 41.
F. Turck,F.Fornara,G.汇编(2008)弗洛登的调节和身份:开花轨迹T移动中心阶段。Annu Rev Plant Biol 59,573-594。https://doi.org/10.1146/annurev.arplant.59.032607.092755.
- 42.
S. D. Michaels, R. M. Amasino (1999)开花基因座C.编码了一个新的MADS结构域蛋白,作为开花的抑制因子。植物细胞11,949 - 956。https://doi.org/10.1105/tpc.11.5.949.
- 43.
G. G. Simpson,P.P. Dijkwel,V. Quesada,I. Henderson,C. Dean(2003)fRNA 3 '末端加工因子是否与之相互作用葬礼控制拟南芥植物的过渡。细胞13,777 - 787。https://doi.org/10.1016/s0092 - 8674 (03) 00425 - 2.
- 44.
D. Z.Wu,Z. Liang,T. Yan,Y. Xu,L. J.Xuan,J. Tang,G.Zhou,U. Lohwasser,S. J. Hua,H. Y.Wang等.(2019)全球油菜籽种质的全基因组重测序揭示了生态型分化的遗传基础。Mol Plant 12,30 - 43。https://doi.org/10.1016/j.molp.2018.11.007.
- 45。
R. Raman, S. Diffey, J. Carling, R. B. Cowley, A. Kilian, D. J. Luckett, H. Raman(2016)澳大利亚粮食产量的定量遗传分析芸苔栗鸟双倍单倍体人口。农作物牧场SCI 67,298-307。https://doi.org/10.1071/Cp15283.
- 46。
冯伟,Michaels SD。双重角色f在规范中方法.植物信号表现。2011; 6:703-5 https://dx.doi.org/10.4161%2FPSB.6.5.15052。
- 47。
J. Wang,C. J.Hopkins,J. N.Hou,X. X. Zou,C.N.Wang,Y.Long,S.Kulup,G. J.Kik,J.L.Meng(2012)Brassicaceae谱系的促进剂变异和转录性偏差开花轨迹T..Plos一个7,E47127。https://doi.org/10.1371/journal.pone.0047127.
- 48.
D. R. Bentley,S. Balasubramanian,H.P.P.Swerdlow,G.P.Smith,J.Milton,C. G. Brown,K。霍尔,D. J.Evers,C.L.Bignell等,H.R.Bignell等.(2008)精确的全人类基因组测序使用可逆终止化学。53至59大自然456年。https://doi.org/10.1038/nature07517.
- 49.
T. Maruki,M. Lynch(2017)基因型从人口基因组测序数据调用。基因基因组遗传群7,1393-1404。https://doi.org/10.1534/g3.117.039008.
- 50。
M. M.Malmberg,D.M.Barbulescu,M.C.C.Dakurest,M.Shinozuka,P.Thakur,Y.O. Ogaji,G.C.Spangenberg,H. D. D. D.One.Cogenberg,N.O.I.Cogan(2018)使用脱脂全基因组在油菜中重新测序进行常规基因分型的评估和建议。前植物SCI 9,1809。https://doi.org/10.3389/fpls.2018.01809.
- 51.
J. S. Brouard, B. Boyle, E. M. Ibeagha-Awemu, N. Bissonnette(2017)牛群体的低深度基因分型测序(GBS):最大化选择高质量基因型和准确性的策略。BMC Genet 18,32。https://doi.org/10.1186/s12863-017-0501-Y..
- 52.
F. Henning, H. J. Lee, P. Franchini, A. Meyer(2014)维多利亚湖慈鲷鱼水平条纹的遗传作图:使用RAD标记进行密集连锁作图的优点和缺陷。Mol Ecol 23, 5224-5240。https://doi.org/10.1111/mec.12860.
- 53.
P.Bajgain,M.N. Rouse,J.A.Anterson(2016)比较逐序列和单核苷酸多态性芯片基因分型对小麦的定量特质基因座映射。作物SCI 56,232-248。https://doi.org/10.2135/cropsci2015.06.0389.
- 54.
W. Hussain, P. S. Baenziger, V. Belamkar, M. J. Guttieri, J. P. Venegas, A. Easterly, A. Sallam, J. Poland(2017)基因分型衍生的高密度连锁图谱及其在面包小麦旗叶性状QTL定位中的应用。Sci Rep 7, 16394。https://doi.org/10.1038/s41598-017-16006-z.
- 55.
C. Saintenac,D. Y. Y.Jiang,S.C.Wang,E.Akhunov(2013)多倍体小麦基因组的序列映射。G3 - 基因基因组。遗传。3,1105-1114。https://doi.org/10.1534/g3.113.005819.
- 56。
余凤琴,张晓刚,彭刚,K. C. Falk, S. E. Strelkov, B. D. Gossen(2017)通过基因分型测序揭示了6种病型对杆状茎病抗性的3个QTLPlasmodiophora Brassicae.在芸苔属植物拉伯.SCI REP 7,4516。https://doi.org/10.1038/s41598-017-04903-2.
- 57。
A. S. Mason, E. E. Higgins, R. J. Snowdon, J. Batley, A. Stein, C. Werner, I. A. Parkin (2017芸苔属植物60K Illumina Infinium SNP基因分型阵列。Theor Appl Genet 130, 621-633。https://doi.org/10.1007/s00122-016-2849-1.
- 58。
N. A. Tinker,S. M. Chao,G. R. Lazo,R.Lazo,R. O. Oliver,Y.Huang,J.A.Pland,E. N.Jellen,P.J.J.Maugh,A. Kilian,E. W. Jackson(2014)用于六倍普通燕麦的SNP基因分型阵列。植物基因组7。https://doi.org/10.3835/plantgenome2014.03.0010.
- 59。
洪洪贤,徐磊,刘军,魏文东,苏志强,宁碧涛,R. Perkins,葛文国,K. Miclaus,张磊等.(2012)用于全基因组关联研究的基因分型SNP序列的技术重现性。PLoS One 7, e44483。https://doi.org/10.1371/journal.pone.0044483.
- 60。
J. C. Richter,C. Mollers(2018)冬季油菜春化要求的遗传变异。Acta Hortic 1202,87-91。https://doi.org/10.17660/ActaHortic.2018.1202.13.
- 61。
M. Rapacz,A. Markowski(1999)冬季耐寒,冻胀和欧洲冬季油菜的挫败要求(芸苔栗鸟var。识别鉴定)的品种。中国农学通报,2018,37(6):733 - 738。https://doi.org/10.1046/j.1439-037x.1999.00346.x.
- 62。
A. Kassambara(2018)'GGPLOT2'的出版物就绪图。访问:2018年3月19日。https://github.com/kassambara/ggpubr..
- 63。
P. E. Bayer, B. Hurgobin, A. A. Golicz, C. K. Chan, Y. Yuan, H. Lee, M. Renton, J.孟,R. Li, Y. Long等.(2017)大会和比较两个密切相关的芸苔栗鸟基因组。植物营养与肥料学报15(3),413 - 418。https://doi.org/https://doi.org/10.1111/pbi.12742.
- 64.
Illumina Inc.(2017年)(2017年)访问:2019年5月7日。http://sapac.support.illumina.com/downloads/bcl2fastq-conversion-software-v2-20.html.
- 65.
Najoshi(2013)军刀。访问:2019年5月7日。https://github.com/najoshi/sabre.
- 66.
Trimmomatic: Illumina序列数据的灵活微调器。生物信息学30,2114 - 2120。https://doi.org/10.1093/bioinformatics/btu170.
- 67.
S. R. Andrews (2010) FastQC:高通量序列数据的质量控制工具。访问日期:2018年9月4日。http://www.bioinformatics.babraham.ac.uk/projects/fastqc.
- 68.
P. Ewels, M. Magnusson, S. Lundin, M. Kaller (2016) MultiQC:在单一报告中总结多种工具和样品的分析结果。生物信息学32岁,3047 - 3048。https://doi.org/https://doi.org/10.1093/bioinformatics/btw354.
- 69。
李H(2013)。用BWA-MEM对序列读取、克隆序列和装配序列进行比对。arXiv预印在https://arxiv.org/abs/1303.3997.
- 70。
H. Li, B. Handsaker, A. Wysoker, T. Fennell, J. Ruan, N. Homer, G. Marth, G. Abecasis, R. Durbin(2009)序列比对/地图格式和SAMtools。生物信息学25,2078 - 2079。https://doi.org/10.1093/bioinformatics/btp352.
- 71。
B. S. Pedersen, A. R. Quinlan (2018) Mosdepth:基因组和外显子的快速覆盖计算。生物信息学34岁,867 - 868。https://doi.org/10.1093/bioinformatics/btx699.
- 72。
A. R. Quinlan,I. M. Hall(2010)Bedtools:灵活的公用事业套件,用于比较基因组特征。生物信息学26,841-842。https://doi.org/10.1093/bioinformatics/btq033.
- 73。
M. A. DePristo, E. Banks, R. Poplin, K. V. Garimella, J. R. Maguire, C. Hartl, A. A. Philippakis, G. del Angel, M. A. Rivas, M. Hanna等.(2011)使用下一代DNA测序数据进行变异发现和基因分型的框架。NAT Genet 43,491-498。https://doi.org/10.1038/ng.806..
- 74。
P. DaneCek,A. Auton,G. Abecasis,C. A. Albers,E. Banks,M. A.剥夺,R. E. Hander,G. Lunter,G.T.T.Marth,S. T. Sherry等.(2011)不同的调用格式和VCFtools。生物信息学27岁,2156 - 2158。https://doi.org/10.1093/bioinformatics/btr330.
- 75.
P. Cingolani, A. Platts, L. L. Wang, M. Coon, T. Nguyen, L. Wang, S. J. Land, X. Y. Lu, D. M. Ruden (2012) A program for annotation and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome .黑腹果蝇菌株W(1118);ISO-2;ISO-3。飞行6,80-92。https://doi.org/10.4161/fly.19695.
- 76.
A. Scheben(2019)访问日期:2019年5月7日https://github.com/ascheben/bn_gbs.
- 77.
B. S. Pedersen, A. R. Quinlan (2017) cyvcf2:使用Python快速、灵活的变体分析。生物信息学33岁,1867 - 1869。https://doi.org/10.1093/bioinformatics/btx057.
- 78.
E. Jones,T. Oliphant,P. Peterson(2001)Scipy:用于Python的开源科学工具。访问:2019年1月19日。http://www.scipy.org/.
- 79.
吴玉华,P. R. Bhat, T. J. Close, S. Lonardi(2008)利用图的最小生成树高效准确地构建遗传连锁图。PLoS Genet 4, e1000212。https://doi.org/10.1371/journal.pgen.1000212.
- 80.
J. Taylor, D. Butler (2017) R package ASMap:高效遗传连锁图谱构建与诊断。J Stat软件79,1-29。https://doi.org/10.18637/jss.v079.i06..
- 81.
丘吉尔、多奇数量性状作图的经验阈值。遗传学。1994;138:963 - 71。
- 82.
RStudio Team (2015) RStudio: Integrated Development for R.访问时间:2017年11月11日。http://www.rstudio.com/.
- 83。
F. Bouche, G. Lobet, P. Tocquin, C. Perilleux (2016拟南芥.核酸RE4,D1167-D1171。https://doi.org/10.1093/nar/gkv1054.
- 84。
P. Lamesch, T. Z. Berardini, d.h. Li, D. Swarbreck, C. Wilks, R. Sasidharan, R. Muller, K. Dreher, D. L. Alexander, M. Garcia-Hernandez等.(2012)拟南芥信息资源(TAIR):改进的基因注释和新工具。核酸RES 40,D1202-D1210。https://doi.org/10.1093/nar/gkr1090.
- 85。
S. F. Altschul, W. Gish, W. Miller, E. W. Myers, D. J. Lipman(1990)基本局部对齐搜索工具。中华医学会生物学及分子生物学分会。https://doi.org/10.1016/s0022 - 2836 (05) 80360 - 2.
- 86。
C. Camacho,G.Coulouris,V.Avagyan,N.Ma.Ma,J.Papadopoulos,K.Bearer,T. L. Madden(2009)Blast Plus:架构和应用。BMC生物信息学10,421。https://doi.org/10.1186/1471-2105-10-421.
- 87。
J. H. Yang,K. Osman,M.Iqbal,D. J. Stekel,Z.W. Luo,S. J. Armstrong,F. C. H. Franklin(2013)推断芸苔属植物拉伯使用蛋白质-蛋白质相互作用数据的相互作用组拟南芥.植物科学3,297。https://doi.org/10.3389/fpls.2012.00297.
资金
这项工作由澳大利亚政府和西澳大利亚政府资助的Pawsey超级计算中心提供的资源支持。A.S.得到了澳大利亚政府授予的知识产权的支持。澳大利亚研究委员会项目(no . LP160100030, no . LP140100537, no . LP130100925);拜耳在2018年将作物科学业务的这项工作移交给巴斯夫。资助机构没有参与研究的设计、数据的收集、分析、数据的解释或手稿的撰写。
作者信息
隶属关系
贡献
DE、JB和SR构思并监督项目。ASE种植植株并进行表型分析。ASE、AP和DP制备测序库。ASE编写了材料和方法的表型和文库准备部分。AS进行了分析并起草了手稿。AS, DE, JB和SR撰写了最终稿,所有作者提供了评论和编辑。所有作者阅读并批准了最终的手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
巴斯夫支持这项工作并聘请了SR。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1
.使用ASMap构建连锁图谱,其中包含从BnSOSR x BnWOSR F衍生的校正和imputed ddRAD标记2人口。表S2.本研究生成的地图的平均标记和连锁图大小。简称:双单倍体(DH)、重组自交系(RIL)、扩增片段长度多态性(AFLP)、芸薹属60k基因分型阵列(Brassica 60k)。表S3.在开花时间和发芽时间的表型变异2绘制种群和亲本。春型BnSOSR亲本和冬型BnWOSR亲本的表型以各亲本的所有重复表示(n= 4,n = 3, respectively).表S4.QTL使用基因组宽的单QTL扫描进行萌芽时间(B)和开花时间(FT)。对于每个QTL,所解释的(PVE)和添加剂效果(AE)所解释的表型方差。表S5.影响C2染色体候选开花时间基因变异的综述。QTL范围为4,345,729 ~ 24,916,709。表S6.对16个早花期和19个晚花期的F基因进行全基因组撇脂测序,得到2号染色体上候选基因的外显子覆盖率2个人。表S7.影响早花期和晚花期候选基因的基因间变异研究综述2个人。表S8.在硅双酶消化分析。fragg:碎片总数;FragDe:不同末端碎片总数;LengthDeFrag:不同末端片段的总长度;deffrag100 - 600: 100个碱基到600个碱基之间不同末端片段总数;LengthDeFrag100-600: 100个碱基到600个碱基之间不同末端片段的碱基总长度;%覆盖率:100个碱基到600个碱基之间的片段覆盖参考基因组的百分比。表S9.在绘图人口和父母中开花时间和萌芽时间的表型数据。表S10.本研究中使用的194个后代和7个家长个人的样本列表。
附加文件2:图S1
.分离扭曲的基因座跨越所有染色体。显著性阈值(p> 0.01)以红色虚线表示。无花果S2。.未校正遗传图和校正遗传图中连锁群大小的比较。校正后的联动组与未校正的联动组集中对齐。S3无花果。.物理(X轴)和遗传(Y轴)分别在MB和CM的所有染色体上的标记位置。Spearmans的等级相关性测试结果显示在每个绘图的左上角。无花果。S4.结果图为出芽时间(左)和开花时间(右)的QTL,分别位于C2位点4,673,904和4,655,461。AA型为BnSOSR, BB型为BnWOSR。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Scheben,A.,Severn-Ellis,A.A.,Patel,D。等等。利用基因型纠错的DDRAD测序与开花时间的联动映射和QTL分析芸苔栗鸟.BMC植物杂志20.546(2020)。https://doi.org/10.1186/s12870-020-02756-y
已收到:
接受:
发表:
关键词
- 芸苔栗鸟
- 双消化限制 - 位点相关的DNA测序
- 开花
- QTL
- 单核苷酸多态性