摘要
背景
最近使用限制性位点相关的DNA测序(Radseq)技术以鉴定北美和东亚东亚的联系绘图的大量单核苷酸多态性(SNP)杨树物种。然而,还需要欧洲白杨的高密度遗传联系地图(P. tremula.作为定量性状位点(qtl)定位和分子标记辅助选择的工具杨树原产于欧洲的物种
结果
我们从两种Aspen父母基因型的十字架中建立了一个混合F1人口,其在其酚类和形态学性状中分歧。我们进行了122个F1后代的Radseq,并使用参考基因组成功识别了两种父母的父母,得到15,732个高质量的SNPp . trichocarpa.利用2055个SNPs构建亲本和亲本连锁图谱。母系连锁图谱由1000个SNPs组成,包含19个连锁群,跨越基因组3054.9 cM,相邻标记间平均距离为3.05 cM。亲本图谱由1055个snp和相同数量的连锁群组成,总长度为3090.56 cM,平均间隔距离为2.93 cM。利用遗传连锁图谱进行一年生苗高度变异的QTL定位。在雄株连锁图谱中,LG5 (96.94 cM)为最显著的QTL (LOD = 5.73),解释了18%的表型变异。
结论
在Aspen和高密度遗传联系地图中构造的15,732个SNPS多态性的组合P. tremula.特定内的交叉将为QTL测绘和鉴定促进欧洲白杨中的标记辅助选择的鉴定基因提供有价值的源。
背景
杨树树种是快速生长的树木,由于其快速生长和木材质量,经得起各种机械和化学处理,在世界范围内受到赞赏。它们对纸浆和造纸工业以及燃料和能源部门特别重要,能在短时间内提供宝贵的原材料。在欧洲,最具抗冻性和生产力杨树适应不肥沃和酸性土壤的物种是白杨(杨树tremulaL.)[1].尽管有其价值和优势,Aspen具有主要的缺点 - 由真菌引起的心脏腐烂的大规模行李箱伤害Phellinus tremulae.(债券)。债券。等,鲍里斯。据报道,生长速度最高的白杨无性系(即天然三倍体)受心脏腐病病原体的影响较小[1那2].因此,俄罗斯白杨育种计划的优先任务是增长率,抗病患者和冬季耐寒性,反过来与酚类特征密切相关[3.].
芽齐平和芽组的时序是重要的自适应特征,代表生存和增长之间的关键权衡[4.].早期芽脸盆或晚芽组增加了霜冻伤害的风险,而在北纬季节性季节特征期间,在其他树木的资源竞争中,太早的芽组可能是不利的[5.].有关适应性和生长相关性状的遗传结构的知识将使最有价值的商业杨树无性系的有效育种成为可能。
多年生木本植物的育种是一个长期的过程,基因组工具的可用性和应用将有明显的好处。几个显著的基因组资源杨树育种是最近才有的,例如,组装的基因组序列p . trichocarpa[6.,一个34k SNP序列,可用于自然居群的基因分型杨树trichocarpa和别的杨树物种(7.],以及该属不同种的一些遗传连锁图(在[8.])。联系地图允许鉴定与有价值的特征相关的QTL,随后是候选基因鉴定和开发基因特异性标记,其应促进在长期多年生植物中的育种杨树.例如,为界特异性交叉的全SIB F1后部开发的链接图P. tremula.xP. Tremuloides.允许识别与白杨中的性别测定相关的QTL [9.].因此,对性别连锁的基因组区域进行了综述,考虑到位于序列一致的基因组区域的基因位点p . trichocarpa.这些基因作为白杨性别决定的候选基因的潜力被生物信息学评估,建议作为一个目标基因Potri.019G047300 (' TOZ19 ')。随后,一种基于pcr的标记被开发出来,用于确定未开花的白杨个体或幼苗的性别[10].
下一代测序(NGS)等技术为获得用于高分辨率连锁图谱构建的全基因组遗传标记提供了新的振奋人心的机会。最近,限制位点相关DNA测序(Restriction-site Associated DNA Sequencing, RADseq)被用于鉴定北美种间杂交F1后代连锁图谱中的大量SNPsP. deltoides.和P. Simonii.2545个优质SNP标记构建了2个亲本特异性连锁图谱,相邻标记间平均距离约为3cm,为杨树QTL定位研究提供了基础[11].使用相同的F1混合人群P. deltoides.×P. Simonii.Mousavi等人[12]提出了将RADseq技术与全基因组测序技术相结合,在杂交林木中构建密集遗传连锁图谱的策略。
到目前为止只有特定于特定的杨树交叉被评估为高分辨率连锁图谱,尽管有些杨树具有巨大分配区域的物种将提供大多样性的表型特征,其与不同物种中观察到的可变性相当的[3.].在欧洲两个本地人杨树物种具有足够广泛的地理分布,以考虑对自然变化的研究:黑色杨杨(p .黑质)及白杨(P. tremula.) [5.].
分布区域P. tremula.包括西欧和东欧、俄罗斯的欧洲部分、克里米亚、高加索、西伯利亚、远东、哈萨克斯坦、中亚、蒙古、中国、朝鲜半岛北部和阿尔及利亚[1].栖息在各种环境条件下,白杨表现出许多形态和生理特征的可变性。为了分析自然表型和遗传变异,瑞典阿斯彭(SwAsp)收集物被建立,以揭示遗传决定因素的基因型变异对适应重要[5.].对白杨种种子收集的研究阐明了Aspen的异常高水平的表型和基因型变异,暴露的高水平(> 0.55)的候选性,如芽组,芽爆和发作的衰弱和发病。值得注意的是,芽组和生长之间的显着相关性[13].一套完整的SNP标记在白杨连锁图谱上的位置已知,将有助于未来识别控制适应性状的基因。
本研究的目的是构建欧洲白杨(P. tremula.),利用RADseq数据生成的一套完整的SNP标记。其中,双摘要RADseq (ddRADseq)方法[14]用于基因分型。与现有的RADseq方法相比,ddRADseq技术早先被报道为一种高效、低成本的新SNP发现和非模式物种基因分型方法[14那15].该连锁图谱可为快速生长、心腐病抗性、木材品质、根系种植能力、物候和抗冬性等林业重要生物学性状变异的数量性状位点(qtl)鉴定提供依据。
方法
映射的人口
为了构建高分辨率联系地图,F1全SIB映射群是从两种Aspen基因型的特定内交叉(P. tremula.).雄树和雌树都生长在圣彼得堡地区的同一地点,彼此之间的距离只有8米。这些亲代树的年龄相当:雌树30-32岁,雄树28-30岁,这是根据采集核心样本估计的年轮数量得出的。雌株高h = 19.8 m,胸径d = 0.27 m。对于雄性树,这些测量值分别为h = 17.6 m和d = 0.23 m。在同一环境下,除物候性状和耐寒性外,两亲本的树形特征基本一致。雌树生殖芽开花时间比雄树晚10天,营养芽开花时间比雄树晚6天。结果表明,雄性亲本枝条霜害严重,而雌性亲本枝条未发生霜害。
控制过境于2016年4月在圣彼得堡州林业技术大学温室进行。新鲜切割的男性和女性白杨的枝条约1米长时间置于10升干净的塑料水壶中。在室温(20°C)在室温(20°C)孵育10天后,为雄性和雌性树枝观察到胰蛋白酶出苗。通过使用从雄性猫蛋白收集的花粉的小刷子进行女性花卉人工授粉进行杂交。
接下来,与授粉的女性柔绒花的树枝袋装以排除随机空气传播的花粉。14天后,女性柔粉生产的小水果分裂,以释放大量的微小的棉籽。将种子立即种植在填充有湿泥炭基材的小塑料容器(12x10x7cm)中。萌发发生在种植后的几天内发生(附加文件1:图S1)。2周后,将秧苗移入单独的容器中,放置在苗圃中(附加文件)1:图S2)。在2016年5月,幼苗达到了约。然后移栽到更大的独立容器中(9x9x11厘米)。到2016年9月,大约1100个容器苗木已经准备就绪。
然后将幼苗股票分为三个部分,以在三种不同环境中建立F1全兄弟的白杨树种植园。2016年9月在圣彼得堡西南70公里(59°16'33“N,30°08'32”e)的田野中种植了192株幼苗。这个位置被归类为潮湿的大陆气候;波罗的海旋转克朗斯的明显调节影响力导致温暖,潮湿,夏季,长,中等冷湿冬。另一套192种幼苗被运送到西西伯利亚,种植在Surgut的田间苗圃(61°15'00“N,73°25'00”E)。这种环境具有欧洲居气氛气候,冬季长而非常寒冷(1月份每日平均温度(20°C)和短而短但相对温暖的夏季。用于建立南方的杂交白杨树种植园俄罗斯(Voronezh,51°40'19“N,39°11'03”e)。使用F1杂种的圣彼得堡人口构建高密度连杆地图。
DNA隔离
从亲本和122株一年生幼苗的干叶中提取DNA。对于每个样品,0.15 g干叶材料在液氮中冷冻,并用Mixer Mill MM 400 (Retsch,德国)研磨。研磨后的样品用新鲜的洗涤缓冲液洗涤:100mm Tris-HCl, pH 8.0;50 mM EDTA;1 M氯化钠;2-mercaptoethanol 1%;1%聚乙烯吡咯烷酮(PVP) (k-30, SABC.) [16].根据制造方案使用DNA外肠-3试剂盒(Syntol,Moscow)进行DNA提取。用基因组DNA清洁和浓缩器试剂盒(Zymo Research)清洁提取的DNA。
ddRAD库的构建和测序
为六个十个双消化的Radseq(Ddradseq)文库构建为两个家长基因型(每个父母的8个图书馆重复)及其根据Plekhanova等人所述的协议的122个后代(每个人1个文库)。[17].每个样本的DNA浓度归一化为10 ng/ul,在37℃下用0.7单位的hindiii.(NEB,美国)在20-ul反应体积。将反应混合物在65℃下孵育20分钟,停止消化。然后将“条形码”适配器连接到由HindIII,允许汇集样本。连接反应在22℃下进行1小时,体积为50 ul,使用1.6单位的T4连接酶(NEB,美国)。在65°C加热10分钟达到T4连接酶失活。然后,每个样品混合10 ul,同时用agcourt AMPure XP试剂盒纯化。第二次限制性消化采用0.7单位的NlaIII(NEB, USA),在37°C下20-ul反应体积维持1小时,随后在65°C下灭活20分钟。第二个“公用”适配器连接到悬垂端NlaIII.将反应混合体积(50 ul)分散到14个等价物中,对14个等价物进行PCR扩增,扩增DNA片段。PCR混合物(50 ul)包含1× HF缓冲液,每个引物25 uM, 12.5 uM dNTPs和1单位Phusion®高保真DNA聚合酶(NEB,美国)。PCR条件为:98℃30 s, 98℃10 s, 65℃30 s, 72℃15 s, 72℃2 min,共14个循环。所有14个PCR反应均用agcourt AMPure XP试剂盒汇集并纯化。利用2100 Bioanalyzer(美国安捷伦技术公司)自动电泳检测文库的浓度和大小分布。在南加州大学Genome and Cytometry Core的Illumina HiSeq2500测序仪上以150个碱基reads进行测序。Illumina的数据可在NCBI生物项目PRJNA395596下获得。
SNP打电话
illumina读被映射到p . trichocarpa参考基因组(6.]使用BWA MEM [18在默认映射参数下。多态被称为使用GATK管道[19]这考虑了indel重新调整和基本质量分数重新校准,并通过Gatk中的HaplotypPaller程序同时呼叫所有样本的变体。根据GATK最佳实践建议使用标准硬滤波参数过滤变体[20.那21].更确切地说,过滤GBS数据只能通过深度(QD)> 24,仅保留具有映射质量(MQ)> 37和质量的SNP呼叫。考虑到映射和基因型调用的质量,以确保只有最高的映射和基因型调用的质量有信心。SNP也被过滤以保留具有MQRANKSUM <| 2.0 |的那些,这确保了两种等位基因的映射质量评分没有差异。这种过滤保留了传递所有三个标准的过滤。最后,vcftools软件[22> 3%的小等位基因频率(MAF), 90%以上的基因型呼叫率,HWE精确P.- 超过10-5.
联系地图建设
采用伪testcross mapping策略构建白杨RADseq连锁图谱[23],通过使两个不相关的高度杂合子亲本杂交产生一组F1后代而形成一个定位群体[15].采用该策略,利用相邻标记间的连锁阶段分别构建了亲本和亲本的连锁图谱。
使用JoinMap 3.0构建了一个链接映射[24].在5.0的赔率(LOD)的对数(LOD)的对数为5.0时,联动组分配。回归映射算法和Kosambi的映射函数用于使用以下设置的地图结构:rec = 0.4,lod = 1.0,跳转= 5.由JoinMap 3.0输出绘制的结果链接映射。
QTL定位
使用Windows QTL Cartographer v.2.5实现复合区间映射(CIM)分析[25[步行速度为2厘米,I型错误率为5%。分析了窗宽10cm的五个背景标记的间隔,以控制QTL背景效果。选择Kosambi映射功能以重新计算基因型数据的地图。LOD意义阈值由具有1000个排列的置换测试确定的显着性水平P.<0.05。使用自定义Perl脚本绘制QTL结果,可根据要求提供。
结果
ddRAD库的构建和测序
以2个亲本(每个亲本8个重复)及其122个F1后代为材料,构建了138个ddRAD文库。对138个ddRAD库进行了测序,生成了23656万个读取,包含大约33.83 Gb测序数据(附加文件2:图S3)。女性和男性父母的数据集分别包含2017万reads(包含2.88 Gb)和1308万reads (1.87 Gb)。从122个后代中,产生了204108万reads,包含29.07 Gb的数据用于SNP检测。82.62%和81.89%的读被成功映射到p . trichocarpa分别为母本和父本的参考基因组。在122个子代中,映射读的平均百分比为77.51%。
SNP基因分型
通过过滤标准,GATK确定了16,234个SNP,进一步分析(参见方法)。使用这些SNP,通过使用掺合软件的最大似然估计来筛选122个后代,通过最大似然估计[26].祖传人群的数量是k = 2,对应于每个女性和男性父母的8次(附加文件3.:图S4)。122个F1后代中有7个祖先不明确,排除在进一步分析之外,而115个后代完全显示出杂种特性,并用于连锁图谱构建。
接下来,对16,234个SNP中的每一个进行Chi-Square试验,以检查122f1后代内的孟钟偏析比对孟德尔分离比率1:1,1:1,1:1:1。那些SNP从孟德尔比率偏离(P.从分析中排除> 0.05)。在2之间没有显示出不同多态性的SNPP. tremula.双亲,但多态性与p . trichocarpa参考。这些SNPs被认为是潜在的有价值的标记,并被纳入了在aspen中用于基因分型目的的15732个健壮SNPs的最终列表(附加文件)4.).
高分辨率遗传图谱构建
构建女性地图snp 15732健壮的snp列表选择如果我)孕产妇杂合的(ab)和父亲的纯合子(aa),(二)显示隔离比1:1的后代,iii)显示清晰的多态性当八套母体基因型和8代表的父亲的基因型进行比较,iii)在整个F1种群中缺失的数据少于10%。根据这些标准,选择1000个snp进行女性图谱构建。与此相对应,通过上述标准,选择1055个父系杂合子(ab)和母系纯合子(aa)的SNPs,构建雄性连锁图谱(表)1).
由于不同的联系地图之间的同步和共同性的高水平杨树物种和物理地图p . trichocarpa,[11那12那27我们根据SNPs在参考基因组的19条染色体上的分布情况,将其划分为19个初步类群p . trichocarpa.利用JoinMap 3.0的默认参数,对19条染色体的雄性和雌性图谱分别进行SNP标记。为了确定染色体上SNP标记的最佳作图顺序和精确估计遗传距离,对某一特定连锁群缺失数据超过10%的F1后代进行作图。因此,每条染色体参与SNP作图的F1个体数量在99 - 115之间。
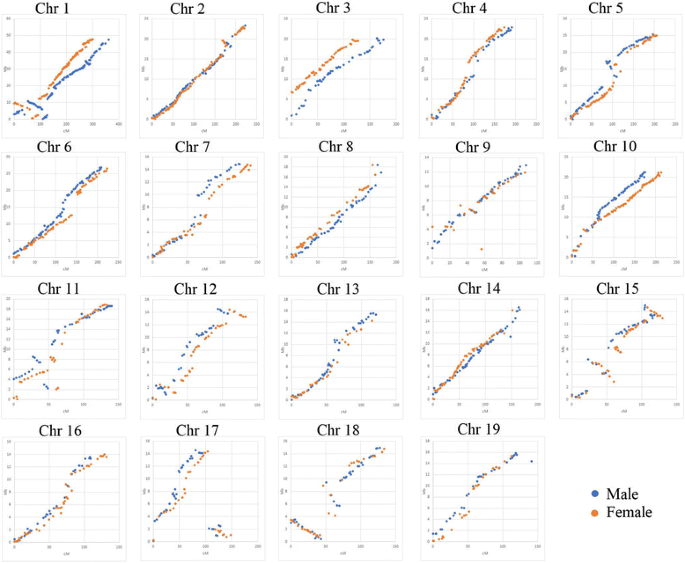
对于所得母体映射1000NP,在10.0的LOD阈值下分配到19个连接基团,其总遗传距离为3054.99cm。单个LG的长度范围为106.01cm(lg9)至300.24cm(lg1),平均分隔距为3.05厘米(表2).对应的父本图谱总长度为3090.56 cM,范围为107.3 cM (LG9) ~ 361.07 cM (LG1),相邻标记间的平均距离为2.93 cM。在19个连锁群的长度上,两个亲本的图谱具有很好的一致性,总相关系数为0.91。LG2和LG9的相似性最高,LG3的不一致性最深。两个亲本连锁图谱如图所示。1和2;相邻SNP标记之间的遗传距离和连锁阶段的详细信息列在附加文件中5.和6..
我们还估计了亲本和亲本的同源关系P. tremula.到的实体地图p . trichocarpa(图。3.).阿斯本父母遗传地图中的SNP标记的顺序和p . trichocarpa基因组高度一致,显示出两个基因组之间高水平的同源性杨树物种。但是,对于一些连杆组例如。CHR 1,11,12,15,17,18(图。3.)检测到一种SNP块,其遗传距离不一致到染色体中的各自的SNP位置P. Trichocarpa。由于母系和父系的图谱显示了相对于参考基因组的相同的不一致性模式,这表明存在染色体倒转,例如在Chr 1,11,15,18上发现的染色体倒转。
最近发表的高分辨率联动地图P. deltoides.和P. Simonii.可以估计物种的19条染色体的遗传长度和物理染色体大小之间的相关性p . trichocarpa[11].在本研究中,我们开发了高密度遗传图P. tremula..因此,估计所有四种染色体尺寸之间的相关性是值得的杨树物种(表3.).遗传图谱间的相关系数最高,为0.98P. tremula.男性父母和物理地图p . trichocarpa,说明该联动图在标记排序方面是相当准确的。雄性连锁群遗传长度的相关系数P. tremula.地图和在地图P. deltoides.和P. Simonii.几乎相同(分别为0.927和0.929)。
利用所构建的高分辨率遗传图谱进行白杨生长相关性状的QTL定位
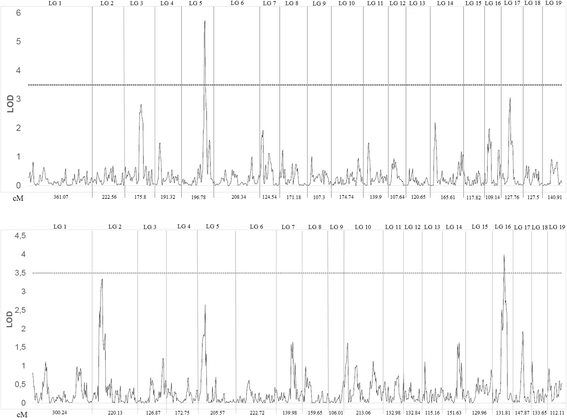
F1映射群体来自特定内的两个十字P. tremula.建立基因型以监测不同环境条件下杨树的表型性状变异。为此,我们在3个不同地理位置建立了F1代的试验种植,以进一步进行QTL定位研究。由于2017年F1群体只有1年的幼苗可用于表型评估,我们估计了幼苗在田间种植前的株高变异(附加文件)7.:图S5)。在作图群体的杂交种中,秧苗高度变化很大,从15厘米到127厘米,服从正态分布(P.= 0.5450)(附加文件7.:图S6)。幼苗高度QTLS的估计的显着阈值使用置换测试确定为LOD = 3.4(P.< 0.05)。最显著的QTL (LOD = 5.73)位于雄性连锁图谱LG5 (96.94 cM)上,解释了18%的表型变异。4.).最近的共分离标记为NC_008471.2_15136737;NC_008471.2_13107624和NC_008471.2_12784748。在SNPS NC_008482.2_6139958和NC_008482.2_6542789之间的间隔,在70.84cm的阴图的LG16上鉴定了与一年幼苗高度变化相关的另一种重要的QTL。其对表型变异的贡献估计为15%。
讨论
在本研究中,我们首先报告了欧洲白杨(P. tremula.).这是最普遍的杨树北半球的物种和港口在人口中和整个物种范围内的遗传多样性。许多核基因的核苷酸多态性水平P. tremula.据报道,比其他木质的、长寿的多年生植物,如松果体和日本柳杉,比那些高出2至5倍桦木属翻车机。值得注意的是,估计的种群细分很低,因此,白杨物种多样性的很大一部分可以从单个种群中取样[28].
由于LD的快速衰减p . tremula以前的假设是,为了确保足够的基因组覆盖以进行QTL定位,需要的标记数量可能过高,在该物种中不可行。另一方面,LD的快速衰减表明,在非常精细的尺度上绘制功能变异是可能的P. tremula.[28].随着NGS技术的发展,克服了标记数量的限制(例如Radseq [29]),所以目前可以采取白杨中低LD的优势,以实现精细的映射和候选基因识别P. tremula..
在白杨中有许多潜在的QTL精细映射的目标。巨大的自然内含性遗传变异P. tremula.表现在表型多样性上,并不断引起森林研究者的关注。至少10P. tremula.在上个世纪只是通过叶子的形状描述了品种(var。Monticola,Parvifolia,Grandifolia,Rotundifolia,未成年人,Outodonta,Stricka,Laevigata,Pendula,仰卧)基于树皮色差报告了两个品种:非常普通的浅色彩色P. tremula.var。vulgaris.和深色的P. tremula.var。Fisca.偶尔发生在俄罗斯的中部和南部地区[30.].物候小种也被指定为种内分类群:晚花蕾开花的白杨(P. tremula.l . var。TardifoliaN. Rubtz.)源自跨伊犁阿拉陶(天山)[31.]并报道白俄罗斯[32.].晚花蕾的白杨比早花蕾的白杨晚10-15天。修正白杨的种内多样性A.Tzarev [3.据报道,俄罗斯森林 - 草原地区(Voronezh地区)的森林 - 草原地区发生了3%的发生。在那里,芽冲洗延迟根据春天的天气条件而变化,随后需要有效温度1.7-2折比白杨的早期形式为1.7-2折。值得注意的是,30岁以上的早期和晚芽冲洗品种的种植园的检查表明,后期腐烂的树木的百分比较低的树木种植园比早期阿斯彭早期的种植园(早期)3.].传统上,特别注意的是具有天然耐心抗性的白杨克隆。从十九世纪开始,在森林中定期在森林中定期描述了健康的白杨树的小型碎片自然群体菲尔丁俄罗斯中部的一种病原体同样,在俄罗斯西北部的白杨森林中,50-60棵被感染的树木在晚期(80-90年)产生了矮林芽,没有任何可见的真菌破坏迹象。我们可以直观地得出这样的结论菲尔丁白杨中的病原体是一种适合育种的遗传性状[1].
在本文中,我们报告了一组15,732个SNP标记,这些标记是两种基因型之间的多态性P. tremula..这只是白杨中的核苷酸多态性的子集,并且不包括这些区域P.tremula没有同源性p . trichocarpa参考基因组。大约20%的读取没有映射到参考,因此,阿斯彭中的一部分多态性仍然仍未探索。映射到的30%的SNPp . trichocarpa参考基因组在本研究中评估的母体和父系树脂树中产生的杂合学基因型。这与前面的报告良好的一致性,显示了29.2%的基因座p . trichocarpa携带杂合基因型[7.].这一组15732个SNP标记将有助于进一步研究白杨的关联图谱。
成功用于建设F1全兄弟姐妹的高分辨率孕产妇和父亲和父系联系地图的两千五十五个SNP标记P. tremula.人口。构建的图谱包含19个连锁群,其中男性图谱为1055个SNPs,女性图谱为1000个SNPs。两个亲本遗传图谱的估计长度分别为3054.99和3090.56 cM,略短于之前报道的高密度图谱的总遗传长度P. deltoides.(4057厘米)P. Simonii.(3.809厘米)(11].另一方面,这些值大于报告的值p . adenopoda(2443.2厘米)P. Alba.(2719.5)[27基于SSR和SRAP标记。在我们的研究中,白杨连锁群的遗传长度与染色体的大小一致p . trichocarpa(表1那2).
尽管在染色体上的SNP序列之间存在高度保守的共线性和共线性P. tremula.和p . trichocarpa,我们的结果表明,在这两个分类遥远的物种之间存在一些染色体倒位杨树物种。P. tremula.属于该部分杨树(syn。le多信),而Balsam poplarsp . trichocarpa和P. Simonii.来自同一部分Tacamahaca.shach。就算是P. Simonii.遗传地图,几个连杆组相对于呈现某些SNP的逆订单p . trichocarpa基因组[11].此外,对于几乎所有的连锁集团P. deltoides.和P. Simonii.一个或多个局部区域的SNP序列已被报道与参考基因组位置不一致[12].我们假设一些联系群体P. tremula.(如Chr 1, 11, 15, 18)染色体倒位可以相对暴露于SNP标记序列上p . trichocarpa染色体。当染色体在两点断裂,由断点所限定的片段以相反的方向重新插入时,就会发生反转[33.].逆转的关键进化效果是它们抑制重组作为杂合子。结果,逆转可能有助于植物中的生殖隔离,局部适应和物种[34.].全基因组测序的比较P. tremula.的参考基因组p . trichocarpa可以确定在两个基因组上确定是否观察到SNP标记的顺序是由于染色体的反转,而不是遗传图谱的变形。
结论
在本研究中,我们描述了一组15732个SNPs的多态性p . tremula一种在所有树种中地理分布最广的植物。采用RADseq基因分型法对2个杨树亲本及其122个F1代进行snp分析,并对2017万个Illumina reads进行图谱分析p . trichocarpa参考基因组。2055个SNP标记成功构建了白杨种内杂交高分辨率的亲本和亲本连锁图谱。利用所构建的连锁图谱,成功地定位了全亲本家族中与株高变异相关的qtl。高分辨率的连锁图谱和SNP标记集将为QTL定位和关联研究提供有价值的资源P. tremula.促进欧洲白杨的标记辅助选择。
参考文献
- 1.
Yablokov。培养和培育健康的白杨。列宁格勒:Goslesbumizdat;1949.在俄罗斯
- 2.
Garipov Nr。三倍体白杨的选择和传播(杨树tremulaL.)在鞑靼斯坦共和国分子遗传学和生物技术方法中的应用。2014年。http://dlib.rsl.ru/010055526669..2017年10月16日访问。
- 3.
杨树的“分类学”。沃罗涅日:沃罗涅日州立大学;1985.在俄罗斯
- 4。
Frewen BE, Chen TH, Howe GT, Davis J, Rohde A, Boerjan W, Bradshaw HD。芽集和芽涌的数量性状位点及候选基因定位杨树.遗传学。2000;154(2):837 - 45。
- 5。
Luquez V,Hall D,Albretsen Br,Karlsson J,Ingvarsson P,Jansson S.(2008)。Aspen的自然候酶变异(杨树tremula): SwAsp集合。植物遗传学报。2008;4(2):279-92。
- 6。
张志强,张志强,张志强,等。黑杨的基因组杨树trichocarpa(托。&灰色)。科学。2006;313:1596 - 604。PMID: 16973872
- 7。
Geraldes A,Difazio Sp,Slavov Gt,Ranjan P,Mandero W,Hannemann J等人。用于Populus Trichocarpa的34k SNP基因分型阵列:设计,应用于自然人群体的研究和对其他的可转移性杨树物种。生态学报。2013;13(2):306-23。
- 8。
王颖,孙旭,谭斌,张斌,徐兰,黄敏,王敏杨树adenopoda的格言。×P. Alba.基于SSR和SRAP标记的L.杂种。Euphytica。2010;173(2):193 - 205。
- 9。
关键词:SSR分子标记,SSR分子标记,连锁群,遗传定位杨树tremula×美洲山杨十字架。acta photonica sinica; 2011;41(2): 245-53。
- 10.
基于pcr的白杨性别鉴定方法。植物医学杂志。2015;17(1):256 - 61。
- 11.
佟超,李海华,王玉霞,欧建,王东,等。构建高密度的联动图杨树三角肌×P. Simonii.使用限制位点相关DNA测序。《公共科学图书馆•综合》。2016;11 (3):e0150692。
- 12.
刘峰,吴杰,李华,史军。新创利用限制性内切位点相关DNA和全基因组测序技术在杨树中的SNP发现和遗传连锁定位BMC基因组学。2016;17(1):656。
- 13.
白杨树的秋季衰老不是由白昼长度引起的。杂志杆菌》2017。DOI:10.1111 / ppl.12593.
- 14。
彼得森BK,Weber Jn,Kay Eh,Fisher Hs,Hoekstra他。双层摘要Radseq:一种廉价的模型和非模型物种中的Novo SNP发现和基因分型的方法。Plos一个。2012; 7(5):E37135。
- 15.
Sun C,Niu Y,Ye X,Dong J,Hu W,Zeng Q等。建设高密度连杆地图和普通话中的性别测定和生长相关基因座的映射(Siniperca Chuatsi.).BMC基因组学。2017;18(1):446。
- 16。
李廷堂,杨杰,陈大川,张小林,唐志生。向日葵成熟叶片基因组DNA制备的优化方法。中国科学(d辑:地球科学)2007;6(4):1064-71。
- 17。
Plekhanova E,Vishnyakova Ma,Bulyntsev S等人。Vavilov的历史体育群体基因组和表型分析揭示了农艺性状环境和基因组岛的影响。科学报告。2017; 7:4816。DOI:10.1038 / s41598-017-05087-5.
- 18。
李H,Durbin R.快速准确的长读与挖洞轮转变换。生物信息学。2010; 26(5):589-95。
- 19。
McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A等人。基因组分析工具包:MapReduce框架,用于分析下一代DNA测序数据。Genome Res。2010; 20(9):1297-303。
- 20。
王志强,王志强,王志强,等。利用下一代DNA测序数据进行变异发现和基因分型的框架。43 Nat麝猫。2011;(5):491 - 8。
- 21.
范德奥韦拉,卡内罗·莫,Hartl C, Poplin R, del Angel G,等。从FastQ数据到高可信度的变体调用:基因组分析工具包最佳实践管道。Curr Protoc Bioinformatics. 2013;11:10。
- 22.
等。不同的调用格式和VCFtools。生物信息学。2011;27(15):2156 - 8。
- 23.
Gratepaglia D,Sederoff R.遗传联系地图桉树茅和桉树urophylla使用伪测试杂交:mapping策略和RAPD标记。遗传学。1994;137(4):1121 - 37。
- 24.
van Ooijen,Johan W,Voorrips Re。“JoinMap®3.0,软件用于计算遗传联系地图。”植物研究国际,Wageningen,荷兰。(2001); 1-51。
- 25.
Wang S, Basten CJ, Zeng Z-B。Windows QTL Cartographer 2.5。北卡罗来纳州立大学统计学系,罗利,北卡罗来纳州。2012.http://statgen.ncsu.edu/qtlcart/WQTLCart.htm.2012年8月1日。
- 26。
亚历山大DH,Novembre J,Lange K.基于快速模型的无关个人祖先估算。Genome Res。2009; 19:1655-64。
- 27。
王Y,张B,太阳X,Tan B,徐洛,黄M等。比较基因组映射Populus Adenopoda,P. Alba,P. Deltoides,P. EuRamericana和P.Trichocarpa..基因类型系统。2011; 86(4):257-68。
- 28。
Ingvarsson PK。核苷酸多态性和欧洲天然群体内部和欧洲天然群体中的核苷酸多态性(杨树tremulaL.,salicaceae.).遗传学。2005;169(2):945 - 53年。
- 29。
Baird Na,Etter Pd,Atwood Ts,Currey MC,Shiver Al,Lewis Za等人。使用测序的RAD标记的快速SNP发现和遗传映射。Plos一个。2008; 3(10):E3376。
- 30。
KUNITSKII B. ASPEN植物和造林特征与其使用说明。St.Petersburg森林学院的年鉴。1888; 2:51-71。
- 31。
Rubtzov倪。关于穿越伊犁阿拉托的一种新型白杨的说明。苏联植物学杂志。1938; 23(1): 23日。
- 32。
Kruglikov GG。在新形式的白杨。林业。1938; 6(12):34-42。
- 33.
染色体倒位如何及为何进化。公共科学图书馆杂志。2010;8 (9):e1000501。DOI:10.1371 / journal.pbio.1000501..
- 34.
Lowry DB,Willis JH。广泛的染色体反转多态性有助于主要的寿命历史过渡,局部适应和生殖隔离。Plos Biol。2010; 8:E1000500。DOI:10.1371 / journal.pbio。1000500..
致谢
我们感谢Georg von Wühlisch博士启动了俄德合作MaRussiA项目。我们感谢Dmitry Danilov博士在建立圣彼得堡地区杂交杨树种植园(Druzhnaja Gorka)期间对实地工作的帮助。
资金
本研究由德国食品和农业部(MaRussiA项目,2015-2016)资助;俄罗斯联邦科学和教育部的37.1521.2014/K。SVN与VIR的合作得到了赞伯格基金会USC拨款的支持。出版费用由MaRussiA项目提供。
数据和材料的可用性
支持数据作为附加文件包括在内。
关于这个补充
本文已作为BMC植物生物学第17卷补充1,2017:来自Plantgen 2017的选定文章。补充剂的全部内容可在线获得//www.cinefiend.com/articles/supplements/volume-17-supplement-1.
作者信息
隶属关系
贡献
AVZ创建了F1人口,建立了全兄弟姐妹的种植园,协调了表型数据的集合。PSU建立了联动地图,执行了同步分析。PSU和MVL准备了Radseq库。MVL制造的表型,进行了QTL分析。PLC分析了Illumina读,使SNP呼叫,进行了混合分析。SVN促成了研究的设计,并授予对Illumina测序的访问。EKP构思了这项研究,并与PLC写了稿件。所有作者阅读并认可的终稿。
通讯作者
道德声明
伦理批准和同意参与
本研究不包含任何需要伦理同意或批准的研究。
同意出版物
不适用。
利益争夺
提交人声明他们没有竞争利益。
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
附加文件1:图S1。
两个白杨亲本基因型人工杂交获得的杂交种子(F1)的萌发。图S2。两周大的F1杂交白杨苗木放置在衬出苗圃。(PPTX 1375 kb)
附加文件2:图S3。
成功映射到的138个已排序的ddRAD库和读的信息p . trichocarpa参考基因组。(PDF 391 KB)
附加文件3:图S4。
122 F1 ASPEN后代的个人祖先通过基于16,234个SNP的最大似然方法估算,伴有掺合软件。(JPEG 1846 KB)
额外的文件4:
用于白杨基因分型的15732个可靠snp列表。(TXT 8857 kb)
额外的文件5:
详细信息的遗传距离和相邻SNP标记之间的连锁阶段构建的母体连锁图谱P. tremula.种内的十字架。(XLSX 427 kb)
额外的文件6:
所构建的父系连锁图谱中相邻SNP标记间的遗传距离和连锁阶段的详细信息P. tremula.种内的十字架。(xlsx, 428kb) (xlsx, 430kb)
附加文件7:图S5。
在田地种植植物之前对一岁幼苗高度变异的评估。图S6。IT的Chi-Square良好的IT测试对幼苗高度值的正常性源自来自的122 f1后代P. tremula.种内的十字架。(PPTX 939 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Zhigunov,A.V.,Ulianich,P.S.,Lebedeva,M.V。et al。欧洲白杨(杨树tremula采用RADseq技术。BMC植物BIOL.17,180(2017)。https://doi.org/10.1186/s12870-017-1127-y.
关键字
- 欧洲阿斯彭
- 种内杂交
- 全兄弟姐妹
- RADseq
- 高分辨率连杆图
- SNP.
- 生长相关的特质
- QTL定位